
Last Updated on Haziran 28, 2025 by EDİTÖR
Kapsamlı bir pan-doku hücresi atlası monte etmek için SCRNA-seq veri kümelerini topladık ve Scanpy aracılığıyla kalite kontrol prosedürlerini gerçekleştirdik48 Araç seti, sonraki bölümlerde ayrıntılı olarak açıklandığı gibi (Genişletilmiş Veri Şekil. 1 ve ek masa 1). Aksi belirtilmedikçe varsayılan parametreler kullanıldı.
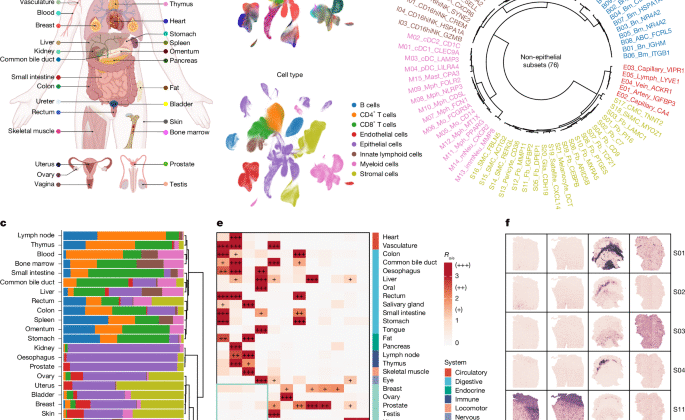
Aşağıdaki kriterleri karşılayan yetişkin örneklerinden SCRNA-seq veri kümelerini dahil ettik: (1) Taze, dondurulmamış örneklerin kullanılması; (2) Hücre tipi zenginleştirmeye dayalı numunelerin dahil edilmesi: (a) hücre tipi zenginleştirme yok; (b) bağışıklık, epitelyal, endotelyal ve stromal bölmelerin bir karışımı; (c) bağışıklık veya immün olmayan hücre popülasyonları için zenginleştirme; ve (3) 10x genomik platformunu kullanarak tek nükleus değil, tek hücreli üretim. Bu kriterler, veri kümeleri boyunca parti efektlerini en aza indirmek için uygulandı49. Nihayetinde, 26 kohorttan toplam 33 veri kümesi dahil edildi ve toplu olarak 35 insan dokusu boyunca bir hücre atlasını temsil etti.
İnsan genom düzeneğinin farklı versiyonlarıyla açıklanmış veri kümelerini standartlaştırmak için, transkriptomu en çok kullanılan 10x genomik gen ek açıklamalarında bulunan ortak 21.812 gen setiyle sınırlandırdık, GRCH38 (Ensembl 84), GRCH38 (Ensembl 93) ve GRCH38 (Gencode V32/ENCLBL 98). Orijinal çalışmalarda düşük kaliteli veya mikrop hattı olarak tanımlanan hücreler hariç tutuldu ve sadece aşağıdaki kriterleri karşılayan hücreler korundu: 500-8.000 gen, 1.000-100.000 gen sayımı ve% 20’den az mitokondriyal gen sayıları. Scrublet uyguladık50Scanpy’ye, her kohorta entegre edilmiş ve tüm kohortlarda 90. persentilin aşan bir çift skoruna sahip çıkarılmış hücreler. Daha sonra 50’den az yüksek kaliteli hücreye sahip örnekleri hariç tuttuk. Sonunda, analiz katı kalite kontrol önlemlerini geçen toplam 700’den fazla örnek içeriyordu.
Tüm veri kümelerinde kombine gen sayısı matrisi ile başlayarak, normalize edilmiş gen ekspresyon matrisini, 10.000 ölçek faktörü ve ardından logaritmik dönüşüm kullanarak hücre başına toplam sayıları (kütüphane boyutu) normalleştirerek türettik. Yüksek değişken genler (HVG’ler) daha sonra aşağıdaki parametrelerle Scanpy.pp.Pp.highly_Variable_Genes işlevi kullanılarak seçildi: (n_top_genes = 2000, flavor = “cell_ranger”, Batch_key = “datasetID”). Özellikle HVG seçimi, immünoglobulin genleri, T hücre reseptör genleri, ribozom protein kodlayan genler, ısı şoku proteinleri ile ilişkili genler ve mitokondriyal genler dahil olmak üzere spesifik genlerin çıkarılmasından sonra gerçekleştirildi. Hücre başına toplam gen sayımları, mitokondriyal gen sayımlarının yüzdesi ve hücre döngüsü dahil olmak üzere birkaç karıştırıcı etki, scanpy.pp.regress_out fonksiyonu kullanılarak ele alınmıştır. Son olarak, HVG’ler tüm hücreler arasında ortalandı ve ölçeklendirildi.
Bu kapsamlı veri kümelerini entegre etmek için, aksi belirtilmedikçe Scanpy araç setini varsayılan parametrelerle kullandık.
Veri kümelerimiz için en iyi entegrasyon yöntemini belirlemek için SCIB’yi kullandık17 Yaygın olarak kullanılan birkaç python tabanlı araçları kıyaslamak için: bbknn18Uyum51Scanorama52 ve derin öğrenme tabanlı SCVI53Scanvi54ve ScalEx55. SCIB’deki 14 metrik arasında, HVG ve yörünge için biyolojik koruma uygulanmamıştır ve 2 TB’lik bellek gereksinimleri nedeniyle Kbet metriği hariç tutulmuştur. Toplam skorlar, toplu düzeltme ve biyolojik varyans korumasının ağırlıklı ortalaması (40/60) olarak hesaplandı. Daha da önemlisi, tüm Atlas ve bir alt küme atlası üzerinde iki bağımsız kıyaslama analizi yaptık. Sonunda, BBKNN en iyi performans sergiledi ve pan-doku veri kümelerinin entegrasyonu için kullanıldı (Genişletilmiş Veri Şekil. 2).
Temel bileşen analizi, 50 temel bileşeni çıkarmak için ortalanmış ve ölçeklendirilmiş HVG ekspresyon matrisi üzerinde gerçekleştirildi. Scanpy’ye entegre olan BBKNN, daha sonra veri kümesi ile toplu değişken olarak yürütüldü. Daha sonra toplu olarak düzeltilmiş grafik, UMAP yapmak için kullanıldı56 İki boyutlu bir düzen üzerindeki hücreleri görselleştirmek için.
En az iki düzey denetimsiz hücre kümeleme ve ek açıklama gerçekleştirdik. İlk kümeleme seviyesi, çözünürlüklü Scanpy.tl.leiden fonksiyonu kullanılarak gerçekleştirildi = 0.1, ardından farklı şekilde eksprese edilen genlerin tanımlanması (DEGS; log;2-Dönüşümlü kat değişiklikleri> 1, fdr <0.05, öğrenci T-test). Sekiz geniş hücre tipi, kanonik belirteçler ve DEG’ler temelinde tanımlanmıştır. Ayrıca CellTypist’ten hücre ek açıklaması konusunda yardım aldık7immune_all_high ve immune_all_low modellerini kullanan otomatik hücre tipi ek açıklama aracı. Daha sonra, her hücre tipi için birkaç farklı hücre alt kümesi elde etmek için bağlama özgü çözünürlükler kullanılarak daha fazla kümeleme (ikinci veya daha fazla seviye) gerçekleştirildi. Epitel hücreleri, yüksek dokuya özgü doğaları nedeniyle daha fazla kümelemeden çıkarıldı. Toplamda, 317 donörde 706 örnekten 2.293.951 yüksek kaliteli hücre, 76 epitelyal olmayan alt kümeye ve 26 epitel hücre tipine açıklandı.
Aynı alt kümeler içindeki tüm hücrelerin gen ekspresyonunu ortalayarak 76 epitel olmayan hücre altkümesi için sahte bulan profiller ürettik. Daha sonra, denetimsiz hiyerarşik kümelenme korelasyon mesafesi ve HCLUST fonksiyonu (Method = “Ward.D”) kullanılarak yapıldı. Sonuçlar DENDEXTEND R paketi kullanılarak görüntülendi.
Birden fazla hücre tipi arasında koordinasyonu sistematik olarak çözmek için tasarlanmış bir hesaplama çerçevesi olan Covarnet’i tanıttık. Covarnet, çeşitli numunelerdeki hücre alt kümesi frekanslarındaki kovaryansı analiz ederek birlikte ortaya çıkan CM ağlarını tanımlar.
Covarnet, her hücre tipi ve numunesi içindeki hücre alt kümesi frekanslarında giriş verilerini kullanır. Birlikte ortaya çıkan alt kümeleri (düğümler) kenarlara bağlayarak CM ağlarını ortaklaşa belirlemek için iki paralel modül kullanır. İlk modül NMF’yi hücre alt kümesi frekans matrisine uygular ve alt kümeleri ağırlıklarına göre önceliklendiren faktörleri tanımlar. Her bir faktörün üst alt kümeleri, tek bir CM ağında birlikte ortaya çıkan düğümler olarak hareket eder. İkinci modül, potansiyel kenarlar olarak işlev gören spesifik olarak korelasyonlu alt küme çiftlerini tanımlar. Daha sonra, bu potansiyel kenarlar aracılığıyla birbirine bağlayan düğümleri birbirine bağlamak için birden fazla CM ağı oluşturulur, ardından topolojik ve istatistiksel değerlendirmeler yapılır.
Hücre-subset frekanslarının dokular ve klinik örnekler arasında karşılaştırılabilirliğini sağlamak için, sadece hücre tipi zenginleştirme olmayan veya dört hücre bölmesinin karışımlarından gelen örnekleri dahil ettik. 50’den az yüksek kaliteli hücreli numuneler hariç tutulmuştur. Her uygun numune için, hücre alt kümelerinin frekanslarını karşılık gelen hücre tipleri içinde hesapladık. Frekans matrisini düzeltmek için min-makx normalizasyonu uygulandı ve farklı hücre alt kümelerinin farklı hücre tiplerinde etkisini hafifletti. Böylece, covarnnet prosedüründe 0 ila 1 arasında değişen düzeltilmiş bir frekans matrisi kullanılmıştır. Özellikle, pan-doku atlası için 76 alt kümeden (satır) ve 510 örnekten (sütunlar) oluşan bir frekans matrisi oluşturduk.
NMF, tek hücrenin analizinde kullanılmıştır57–58–59–60 ve mekansal61–62 gen ekspresyon programlarını çıkarmak için ifade verileri. Bu çalışmada, covarnet, NMF R paketinde uygulandığı gibi, NSNMF yöntemini 2 ila 20 arası rütbelerle kullanan hücresel birlikte ortaya çıkan programları deşifre etmek için frekans matrisine NMF uygular.63. Sağlamlığı sağlamak için, bir fikir birliği çıktısı elde etmek için 30 çalışma yaptık, K her örnekte faktörler ve faaliyetleri. Spesifik olarak, her bir faktörün ilk on alt kümesi, pan-doku analizi için tek bir cm ağında birlikte ortaya çıkan düğüm adayları olarak kullanılmıştır.
NMF analizi için optimal sıralamayı belirlemek için, önceki raporlardan gelen uygulamalara uygun olarak, değerlendirme endeksi olarak copenetik korelasyon katsayısını (CCC) kullandık.1. CCC, 0 ila 1 ve 1 arasında değişen değerler ile sınıflandırma stabilitesini ölçmek için kullanılır.64. CCC’yi sırada gösterdik K gibi RK ve aşağıdaki kriterlere dayalı tutarlı istikrar için bu bağlamda uyarlanmış bir prosedür oluşturuldu: (1) RK – 2< RK – 1 < RK; (2) RK> RK+ 1 . Bu kriterleri karşılayan bir dizi rütbe arasında, optimal rütbe daha sonra CCC’nin maksimize edildiği olarak tanımlandı. Pan-doku atlası için seçilen optimal sıralama 12 idi (Genişletilmiş Veri Şekil. 3a, b).
Covarnet, herhangi bir iki hücre alt kümesinin birlikte olup olmadığını değerlendirmek için Pearson korelasyon katsayılarını kullanır. Belirli bir set için SHücre alt kümeleri, çift korelasyon testleri frekans matrisine göre yapılır ve bu da birS× S korelasyon katsayısı matrisi (gösterilir R ). Korelasyonların özgüllüğünü ölçmek için bir gösterge tanımlanır. Her öğe içinRIJ ( Ben< J) içinde Rarka plan setiSIJ ve özgüllük dizin spesifikasyonu ( RIJ) şu şekilde tanımlanır:
$$ {s} _ {ij} = {{r} {{ik} | K no i } cup {{r} _ {kj} | k no j } $$
$$ { rm {spec}} ({r} _ {ij}) = frac {| {r in {s} _ {ij} | r le {r} _ {ij} } | } {| {S} _ {ij} | } $$
Başka bir deyişle, özgüllük endeksi, arka plan kümesindeki öğelerin fraksiyonu olarak tanımlanır.RIJ. Özgüllük kesimi otomatik bir yöntemle belirlenir. Eğer NVe N Her cm’de varsayılan alt kümeyi ve toplam alt kümeyi, daha sonra özgüllük kesimini temsil eder ( N– N ) şu şekilde belirlenecektir:
$$ { rm {c}} { rm {u}} { rm {t}} { rm {o}} {f}}
Bu yaklaşım, CMS içindeki alt kümelerin ve bunların birlikte ortaya çıkmasının dengeli bir değerlendirmesini sağlar. Spesifik olarak ilişkili alt küme çiftleri korelasyon (katsayı ve FDR) ve özgüllük ile birlikte belirlenir. Pan-doku atlası için 147 çift ürettik. Bu çiftler küresel bir ağ olarak görselleştirildi (Ek Şek. 4).
Her NMF faktörü için, üst alt kümeler potansiyel düğümler olarak belirlenir ve kenarlar, izole düğümleri çıkararak spesifik olarak korelasyonlu alt küme çiftlerini bağlar. Yapılan her CM ağında, bağlantı skoru, gözlemlenen kenarların o ağdaki tüm düğümler arasındaki toplam kenarlara oranı olarak hesaplanır. Bu puanın istatistiksel önemi bir permütasyon testi kullanılarak değerlendirilir ( N= 10.000) düğüm etiketlerinde. IGRAPH R paketini, CM ağlarını görselleştirmek için kullandık, düğümler renk ile kodlanmış düğümler ve spesifikliği yansıtacak şekilde ölçeklendirilmiş kenar renk gradyanları.
Bireysel numunelerdeki CM aktiviteleri, NMF prosedüründen katsayı matrisi ile ölçülür ve tüm CM’lerin her bir numune için 1 eşittir. Her numuneye en bol CM’ye dayanan bir CMT etiketi atandı. Örneğin, bir numune tüm CM’ler arasında en yüksek CM01 aktivitesini sergilediyse, CMT01 olarak etiketlendi. Dokular arasında kullanılan tüm sağlıklı tek hücreli örnekler 12 CMT grubuna sınıflandırılmıştır (Genişletilmiş Veri Şekil. 3e).
Gtex’i kullandık24 Tek hücreli verilerle tanımlanan CMS’yi doğrulamak için RNA-seq veri kümeleri (Genişletilmiş Veri Şekil. 4).
GTEX portalından 17.382 yığın RNA-seq numunesi için milyon (TPM) ve meta veriler başına gen transkriptlerini aldık (V8 salınımı)65. Hücre çizgilerinden türetilen numuneler hariç tutuldu ve ‘serviks uteri’ kategorisi tutarlılık için ‘uterus’ kategorisine birleştirildi. Tutarlılığı sağlamak için, sadece tek hücreli kohortta temsil edilen dokular tutuldu ve daha fazla analiz için 23 doku kapsayan toplam 12.240 örneğe daraldı. Gen ekspresyon verileri, tek tip bir kütüphane boyutuna 10.000 olarak yeniden normalleştirildi ve tek hücreli verilerle karşılaştırılabilirlik için log-dönüştürüldü.
Psödo-top CMT örnekleri arasında DEG’leri tanımlayarak başladık. Kat değişimine göre sıralanan ilk on derece, CMT imza genleri olarak adlandırıldı. Seurat R paketini kullanıyor66bu CMT imza setlerine dayanarak bireysel RNA-seq örnekleri için puanları hesaplamak için AddModulescore işlevini uyguladık. Tüm negatif skorlar sıfıra ayarlandı ve en yüksek skoru 0,2’den az olan numunelerin% 2.3’ü (12.240 üzerinden 278) sağlam sınıflandırma sağlamak için hariç tutuldu. Nihayetinde, geri kalan 11.962 numune, analiz edilen dokular arasında CM temsilinin ayrıntılı bir incelemesini kolaylaştıran 12 farklı CMT olarak kategorize edildi.
Dokular arasında hücre alt kümelerinin prevalansını değerlendirmek için, gözlemlenen (O) her bir alt küme doku kombinasyonu için beklenen (e) hücre sayılarını karşılaştırdık, RO/E= Belirlenen yöntemleri takip eden/beklenen/beklenen35–38–39. Her bir alt küme-doku kombinasyonu için beklenen hücre sayıları ki-kare testinden türetildi ve zenginleştirme olarak tanımlandı RO/E> 1 (Şek. 1e ve Ek Şek. 2). Her CM’nin değerlendirilmesi için, aktivitesini her bir doku içindeki tüm numunelerde ortalama olarak doku seviyesi CM aktivitelerini hesapladık. . RO/EOran, CM profillerinin doku dağılımını gösterdi (Şek. 2C). Toplu ve tek hücreli analizler arasındaki örtüşen 23 dokuda CM zenginleştirmesini karşılaştırmak için bağımsız olarak hesapladık RO/EHer veri türü için oranlar ve karşılaştırma için bunları birleştirdi (Genişletilmiş Veri Şekil. 4F). Sonuçlar Complexheatmap R paketi kullanılarak görüntülendi67.
Çeşitli insan dokuları ve kanser türlerinin yayında mekansal olarak çözülmüş transkriptomik veri kümelerini (Visium ve Xenium) topladık. Bu veri kümeleri için ayrıntılı katılım numaraları ve referanslar sağlanmıştır (Ek Tablo 3).
Mekansal transkriptomik verileri dekonvoluting için, hücre2lokasyonunu kullandık25Mekansal veriler içinde ince taneli hücre tiplerini doğru bir şekilde çözebilen bir Bayes model. Hem sağlıklı hem de kanser veri kümelerini kullanarak, hücre tipi imzalar elde etmek için karşılık gelen entegre SCRNA-seq verilerini referans olarak kullandık. Bu işlemden önce, SCRNA-seq verileri hücre alt kümesi başına 1.000 hücreye alt örneklendi. Hücre tiplerinin 1000’den az hücreden daha az olduğu durumlarda, mevcut tüm hücreler dahil edildi. Cell2location’un önerilen yönergelerine göre ayarladık N= 5 nokta başına beklenen hücre bolluğu ve Ay= 20 RNA algılama duyarlılığında deneyim içi varyasyonu düzenli hale getirmek. Çıktı, her noktada hücre alt kümesi başına beklenen hücre bolluğunu verdi.
Mekansal transkriptomiklerde CMS’yi ölçmek ve görselleştirmek için, NMF sonuçlarından türetilen ağırlıkları uygulayarak her CM içindeki bileşen hücre alt kümelerinin bolluğunu topladık. Ortaya çıkan CM aktiviteleri daha sonra, tüm CM’lerde 99. persentil değeri 1’e ayarlanmış ve nispi büyüklüklerinin doğrudan karşılaştırılmasına izin veren tek tip bir standarda ölçeklendirildi.
Visium noktaları boyunca CMS içindeki hücre-subset bileşenlerinin kolokalizasyonunu değerlendirmek için, bireysel uzamsal bölümler için kolokalizasyon skorunu hesapladık. Her cm için, bu CM içinde en az bir alt kümenin bulunduğu alt küme çiftleri arasındaki Spearman korelasyon katsayılarını hesapladık, bu da bir dizi korelasyon katsayısı ile sonuçlandı. S . CM içindeki medyan korelasyon katsayısı olarak adlandırılır R. Her cm için kolokalizasyon skoru, korelasyonların oranı olarak tanımlanır.Sdaha az veya eşit olan Rküresel bağlamlara göre bir kolokalizasyon ölçüsü sağlar.
Mekansal transkriptomiklerde CMS içindeki hücre-subset bileşenlerinin bölgesel toplanmasını değerlendirmek için küresel iki değişkenli Moran’ın BEN68 SPDEP R paketini kullanarak. Kolokalizasyon skoruna benzer şekilde, toplama skorları Global Moran’ın kullanılarak hesaplandı. BENkorelasyon katsayısı yerine.
Belirlenen CMS için dik olarak doğrulama sağlamak için CellCharter kullandık26 Hücresel nişleri tanımlamak için, mekansal olarak bilgilendirilmiş niş kategorizasyonunu sağlamak için hem gen ekspresyonuna hem de mekansal bilgilere dayanan visium lekelerini kümeleme. Bu analiz, her bir numune için bağımsız olarak, parti etkilerini azaltmak için yapıldı ve aynı dokulardan gelen numuneler arasında sonuçların çapraz doğrulanmasına izin verildi.
Ksenyum platformu, ultra kesin tek hücreli mekansal görüntülemeye sahip hücrelerde ve dokularda yüzlerce ila binlerce genin yerinde karakterizasyonunu sağlar. Yayınlanmış bağırsak ksenyum verilerini kullanarak, CM02 ve CM03’ün mekansal yerlerini karakterize ettik. İlk olarak bu CMS içindeki birden fazla hücre alt kümesini ayırt etmek için bir gen paneli tasarladık, gen transkript yoğunluğu CM yoğunluğu için bir proxy olarak hizmet ediyor (Genişletilmiş Veri Şekil. 5i). Dokunun uzamsal bölgeleri (epitel veya lamina propria)K-Means kümelenmesi ( K= 2) Orijinal veri kümesinde. Doku alt bölgeleri boyunca CM dağılımını değerlendirmek için, bağırsak mukozasında altı farklı alan seçtik ve CM yoğunluklarını ölçtük ve CMS’nin mekansal olarak çözülmüş, tek hücre çözünürlük dağılımını sağladık.
CMS içinde ve genelinde karmaşık hücresel karışıklıkları çözmek için, CellphonedB Python Paketi ile tek hücreli veriler kullanarak ligand-reseptör aracılı hücre-hücre iletişim analizi gerçekleştirdik11–27.
Çok sayıda hücre göz önüne alındığında, her hücre alt kümesinin katkısını eşitlemek için alt örnekleme yaptık. Spesifik olarak, her hücre alt kümesi için hücre sayısını 1.000 hücreye alt örnekledik. Bununla birlikte, belirli alt kümeler için toplam hücre sayısı 1.000’i geçmezse, tüm hücreler analize dahil edildi. Bu yaklaşım, boş dağılımın tüm hücre alt kümelerini doğru bir şekilde temsil etmesini ve daha büyük hücre sayılarına sahip hücre alt kümelerine karşı önyargıdan kaçınmasını amaçlamıştır. Daha sonra, hücre -hücre etkileşimi spesifisitesinin istatistiksel çıkarım için hücre -hücre etkileşimi sayılarına izin veren hücre -hücre etkileşimi spesifisitesinin istatistiksel çıkarımında kullanıldı (genişletilmiş veri Şekil. 6c) veya CMS (Genişletilmiş Veri Şekil. 6d) karşılık gelen hücre alt kümelerinin sonuçlarının ortalaması alınarak. Bu sonuçları alternatif bir araç olan CellChat kullanarak da doğruladık69 (Genişletilmiş Veri Şekil. 6e).
Doku mikroçevreslerinin (CMS) CMS içindeki hücresel karışma üzerindeki etkisini araştırmak için, sırasıyla yüksek veya düşük CM aktiviteleri olan iki numune grubu kullanarak CellphonEDB analizi yaptık. Her cm için, ‘yüksek’ grup, CM’nin tüm CM’ler arasında en yüksek aktiviteyi gösterdiği tüm numuneleri içeriyordu, diğer numuneler ‘düşük’ grup olarak kullanıldı. ‘Örneklerin CMT sınıflandırmalarında’ daha önce tanımlanmış CMT’lerin ardından CM01’i örnek olarak alarak, CMT01 olarak etiketlenmiş tüm numuneler yüksek grup olarak kullanılırken, diğer numuneler düşük grup olarak kullanılmıştır. Daha sonra, iki grup arasındaki karşılaştırmalı analiz, her bir CM’nin bileşenleri olan hücre alt kümelerine odaklanmıştır (Şekil. 3G).
Yakın zamanda bildirilen bağışıklık sözlüğü, 86 sitokine yönelik hücre tipine özgü yanıtlara kapsamlı bir genel bakış sağlar28. Bu temel bilgiyi kullanarak, sitokin yanıtları yoluyla CMS içindeki hücreler arası karışıklığı araştırdık. CM07 ve CM10, bağışıklık hücre alt kümeleri olmadığı için hariç tutulurken, CM12’nin önemli bir sitokin çıkışı yoktu.
Doku mikroçevreleri, hücre-subset fenotipleri üzerinde CM’ye bağımlı bir şekilde geniş bir etki yarattı. Özellikle, her bağışıklık alt kümesi için DEG’lerini belirledik (log2-Dönüşümlü kat değişiklikleri> Günlük2(1.2), FDR <0.05, öğrenci T-test) Scanpy araç setini kullanarak, diğer CMT’lerden gelenlerle karşılaştırıldığında karşılık gelen CMT’lerden numunelerde. Bu tanımlanan DEG’ler, CMS ile ilişkili doku mikro ortamları içindeki sitokinlere yanıt olarak yorumlandı.
İlk olarak, bağışıklık sözlüğü yayınından Ek Tablo3’ü kullanarak her hücre tipi için sitokin imzalarının bir veritabanı oluşturduk28. Dikkat çekici bir şekilde, fare genom bilişim (MGI) veritabanına dayanan fare ve insan ortolog genlerinin bire bir dönüşümünü gerçekleştirdik.70 (http://www.informatics.jax.org/downloads/reports/hmd_humanphenotype.rpt). Daha sonra, Hipergeometrik Testi (FDR <0.05) kullanarak ClusterProfiler R Paketindeki zenginleştirici fonksiyonu ile hücre tipine duyarlı bağışıklık tepkisi zenginleştirme analizi gerçekleştirdik.71.
Sitokin aracılı çok hücreli regülasyonu görselleştirmek için, hem sitokin üretimini hem de yanıtı CM’ye özgü bir şekilde dikkate alarak sitokin ağları inşa ettik. CM’de dikkate alınması için sitokinler için, yanıt vermeyen hücre alt kümelerinden en az birinde 0.1’i aşması gereken sitokin geninin normalleştirilmiş ekspresyon değeri. İki alt birim olan heteromerik sitokinler veya sitokin kompleksleri durumunda, her bir alt birim ayrı ayrı temsil edildi. Igraph R paketi, sitokin ağlarının görsel temsillerini oluşturmak için kullanıldı.
İlk olarak CM aktiviteleri ve dokular arasındaki ilişkiyi, beşten az örneğe sahip dokular hariç inceledik. Her cm için, ayarlanmış doğrusal bir model yerleştirdik R2 açıklanan varyans oranını gösterir. FDR FSonuçların sağlamlığını sağlamak için test bildirildi. CMS’nin güçlü doku tercihleri göz önüne alındığında, sonraki analizler dokuya özgü ilişkilere odaklanmıştır.
Ürünsüz dokularda, CM aktiviteleri erkek ve kadın katılımcılardan alınan örnekler arasında FDR ile ayarlanmış önemi ile karşılaştırıldı. Yaş bilgisi olmayan örnekler analizden çıkarılmıştır. Yaş verileri gruplar halinde kategorize edildi: <35, 35-39, 40-49, 50-59, 60-69 ve 70-85 yıl. 100'den fazla numuneli göğüs veri kümesi (D03) için (Ek Tablo 1), yaş <50 veya ≥50 yıl olarak kategorize edildi. CM faaliyetleri ve yaş grupları arasındaki ilişkiler, istatistiksel anlamlılık ayarlamaları ile değerlendirildi. Spesifik olarak, bağışıklık hücre-subset frekansları ile dalaktaki yaş grupları arasındaki ilişkileri de inceledik.
CMS ve spesifik fenotipik faktörler arasında daha fazla ilişki analizleri gerçekleştirdik. CM09’u lenf nodudaki alkol tüketimi, akciğerde çocukluk tüberkülozu ile CM06 ve CM11, memede menopoz ile CM12 ve uterusta adet döngü fazları ile CM07 ile analiz ettik.
Pizenik kullandık30–72 CM05 içindeki dört alt kümenin (B03, B05, CD4T03 ve I06) regülonlarını çıkarmaya yönelik boru hattı, analizi üç hücresel soy (C hücreleri, CD4 için ayrı ayrı gerçekleştirerek+ T hücreleri ve doğuştan gelen lenfoid hücreler). Her alt küme için regülonlar, regülon özgüllük skorları (RSS) temelinde sıralandı ve her hücre alt kümesi için en yüksek RSS’ye sahip ilk 50 regülon seçildi. Dört alt kümeler arasında on yedi regulon paylaşıldı.
Paylaşılan regülonlar için numune düzeyi aktiviteleri, her numunedeki tüm hücreler arasında hücresel aktivitelerin ortalaması alınarak belirlenmiştir. Daha sonra her regülonun ortalama aktivitesi yaş tabakalı numune grupları arasında karşılaştırıldı. Paylaşılan regülonların hedef genleri soylar arasında karşılaştırıldı ve ilişkilerini göstermek için bir düzenleyici ağ oluşturuldu.
Aynı CMS içindeki farklı hücre tipleri benzer doku mikroçevrelerine maruz kalma eğiliminde olduğundan, koordineli yanıtlar sergileyebileceklerini varsaydık. Bunu araştırmak için diyalog adı verilen bir yöntem kullandık3 CMS için MCP’leri haritalamak için. Bu prosedür parametrenin ayarlanmasını içeriyordu ( K= 3) ve tanımlanan MCP’ler ile diğer fenotipler arasındaki ilişkinin değerlendirilmesi. Bu analiz meme ve CM08’deki CM12’ye uygulandı ve sonuçlanan MCP’ler CM12 programı ve CM08 programı olarak adlandırıldı.
CM08 programını ve inflamatuar ve sitotoksik imzaları karşılaştırmak için, daha önce tarif edildiği gibi dış RNA-seq verilerindeki genel ifadelerini hesapladık.73–74. Özellikle, anti-PD-1 terapilerini takiben ileri melanomlu bireylerden gelen örneklerin RNA-seq veri kümeleri75 indirildi https://github.com/parkerici/morrison-1-public. İmmün hücrelerin inflamatuar imza genleri CD3D– IDO1– Ciita– CD3E– CCL5– Gzmk– CD2– HLA-DRA – CXCL13– IL2RG– NKG7– .– CXCR6– LAG3– Tagap– CXCL10– Stat1Ve GZMB76. Sitotoksik imza genleri IFNG– Gzma– GZMB– PRF1– Gzmk– ZAP70– Gıcırdatmak– Faslg– TBX21– Eomes– CD8A– CD8– CXCL9– CXCL10– CXCL11– CX3CL1– CCL3– CCL4– CX3CR1– CCL5– CXCR3– NKG7– CD160– CD244– NCR1– KLRC2– KLRK1– CD226– Gzz– ITK– CD3D– CD3E– CD3G – Trac – TRBC1– TRBC2– CD28– CD5– Kirdl4– FGFBP2– KLRF1– Sh2d1bVe NCR3(Ref. 77).
CM12 içindeki bağışıklık ve fibroblast alt kümeleri için numune seviyesi enflamatuar skorlarını hesapladık. Fibroblastlar ve bağışıklık alt kümeleri, karşılık gelen enflamatuar gen setleri kullanılarak skorlandı. Fibroblastların inflamatuar imza genleri Mutlu– CHI3L1– MMP3– IL1R1 – IL13ra2 – TNFSF11– MMP10– OSMR– IL11– STRA6– Fap– Wnt2– Twist1VeIL24(Ref. 78). İmmün hücrelerin inflamatuar imza genleri yukarıdaki gibi tanımlanır. Spesifik olarak, ilk olarak her bir numune için tek tek alt kümelerin tüm hücreleri arasındaki ortalama gen ekspresyonunu hesapladık. Daha sonra, Aucell R paketi kullandık30 Numune seviyesi inflamatuar skorları hesaplamak için.
Memede menopozal bir yörünge inşa ettik (veri kümesi D03; Ek Tablo 1) CM12 içindeki hücre alt kümelerinin frekanslarına dayanarak, son bir çalışmada açıklanan metodolojiyi takip ederek5. Hücre alt kümeleri arasındaki frekans farklılıklarının etkisini azaltmak için uyguladıkzıplaFrekans matrisini düzeltmek için normalleştirme. Daha sonra,K-NeighBourhood ve Scanpy.pp.Neighbours ve Scanpy.tl.leiden işlevini kullanarak varsayılan parametrelerle göğüs örnekleri için kümeleme gerçekleştirdi. Menopoz boyunca yörüngeleri modellemek için phate yaptık79 ileA= 40, ardından Palantir kullanılarak psödotim analizi80 standart boru hattı. Başlangıç noktası, yüksek oranda menopoz öncesi örneklere sahip küme olarak tanımlandı.
Reed veri kümesi81 menopozal yörüngeyi doğrulamak için kullanıldı. Veri kümesindeki epitel ile zenginleştirilmiş (‘organoid’) örnekler için, hücre alt kümesi ek açıklaması hücre tipi ile yapıldı7meme dokusunun daha önce açıklanmış gen ekspresyon profillerini referans olarak kullanma. Hücre alt kümesi frekans matrisi daha sonra yukarıda tarif edildiği gibi yörünge analizine girildi.
Malign ilerleme boyunca CMS’nin yeniden kablosunu çözmek için, tek hücreli çözünürlükte bir Pan-kanser transkriptomik atlası oluşturduk (Şekil. 5a).
Sağlıklı veri kümeleri için belirlenen kriterleri takiben, 10x genomik tek hücreli (tek nükleus değil) platformlar kullanılarak üretilen hücre tipi zenginleştirme olmadan taze (dondurulmamış) numunelerden SCRNA-seq veri kümelerini seçici olarak dahil ettik. Bir istisna, numunelerin bağışıklık, epitelyal, endotelyal ve stromal bölmelerin bir karışımını temsil ettiği ESCC_GSE160269 kohortudur. Kalite kontrolü ve diğer ön işlem prosedürleri de sağlıklı veri kümeleri ile tutarlı bir şekilde uygulandı. Toplamda, 48 veri kümesinden 1000’den fazla örnek dahil edildi ve 29 insan majör kanseri tipinde toplu olarak bir hücre atlası oluşturuldu (Genişletilmiş Veri Şekil. 10 ve ek masa 7).
Sağlıklı veri kümelerine uygulanan metodolojinin ardından BBKNN kullanarak veri kümesi entegrasyonu gerçekleştirdik. Tüm hücreler için denetimsiz kümeleme, 0.1 çözünürlüğe sahip Scanpy.tl.leiden fonksiyonu kullanılarak gerçekleştirildi. Daha sonra, kanonik belirteçlere dayalı sekiz geniş hücre tipini belirledik.
Kanserli numunelerde hücre kimliklerini doğru bir şekilde belirlemek için, hücre alt kümesi ek açıklaması için transfer-öğrenme tabanlı bir strateji kullandık. Başlangıçta, sağlıklı verilerde tanımlanan 76 epitelyal olmayan hücre altkümesini kapsayan tek hücreli bir referans veri kümesi ve çeşitli kanser tiplerinde tanımlanan 15 kanserle ilişkili alt kümeyi kapsadık.35–36–37–38–39 (Ek Tablo 9). Toplam hücre sayısı 1.000’i geçmediği sürece, her bir alt kümenin hücre sayısı 1.000’e alt örneklendi. Daha sonra, referans veri kümesi üzerinde transformatör tabanlı bir referans modeli eğitildi. Bunu takiben, pan-kanser atlasından epitel olmayan hücreler referans modeli kullanılarak açıklandı. Bu prosedürler, yorumlanabilir hücre tipi ek açıklama sağlayan çok avlu bir kendine dikkat modeli olan Tosica kullanılarak yürütüldü.82. Tahmin için çağ numarası 15 olarak seçildi (Ek Şek. 8c). Tahmini olasılıkları <0.5 olan hücreler çıkarıldı (Ek Şek. 8d). Sonunda, 717 donörden oluşan 1.062 örnekten toplam 3.038.535 yüksek kaliteli hücre, 91 epitel olmayan alt küme olacak şekilde iyi açıklanmıştır.
Tümör ilerlemesi sırasında çok hücreli dinamikleri karşılaştırmak için, her durumdan en az üç örneği olan sekiz kanser tipi (sağlıklı, bitişik tümör ve tümör) dahil ettik.
Farklı koşullarda sağlıklı CM’leri ölçmek için, her cm içindeki bileşen hücre alt kümelerinin bolluğunu topladık ve NMF sonuçlarından türetilen ağırlıkları uyguladık. Ortaya çıkan CM aktiviteleri daha sonra 0 ila 1 arasında yeniden ölçeklendirildi. Her kanser tipi için sadece en baskın CM düşünülmüştür.
Her kanser tipi için, covarnetin korelasyon analizi modülünü kullanarak spesifik olarak korelasyonlu hücre-subset çiftlerini türettik. Pan dokusu veya pan-kanser analizi ile karşılaştırıldığında, aşağıdaki daha katı kesimler kullanılır: katsayılar> 0.5, FDR 0.95. Sekiz kanser tipinde karşılaştırma yapmak için sadece tanımlanmış özel korelasyonlu alt küme çiftleri kullanılır.
Özellikle, Pan-Cancer Atlas için 91 alt kümeden ve 955 örnekten oluşan bir frekans matrisi oluşturduk. Spesifik olarak, her bir faktörün en iyi 15 alt kümesi, pan-kanser analizi için tek bir cm ağında birlikte ortaya çıkan düğüm adayları olarak kullanılmıştır.
Farklı koşullarda CCM’leri ölçmek için, sağlıklı CMS ile aynı prosedürü kullanarak CCM02 aktivitelerini ölçtük.
Tümör ilerlemesi sırasında CCM02’nin dinamiklerini keşfetmek için, düğümlerin CCM02’de orijinal düğümler olarak uyumlu olduğu iki birlikte ortaya çıkan ağ oluşturduk, kenarlar ise iki senaryoda yeniden hesaplandı. Birinde sağlıklı ve bitişik tümör olmayan örnekler, diğeri ise tümör ve bitişik tümör olmayan örnekler kullandı. Böylece, CM ağları değişmemiş düğümler ve yeni kenarlar kullanılarak oluşturuldu.
CCM02’nin her hücre alt seti bileşeni için, DEG’lerini tanımladık (log2-Dönüşümlü kat değişiklikleri> Günlük2(1.2), FDR <0.05, öğrenciT-test) tümör örneklerinde bitişik tümör olmayan numunelerle karşılaştırıldığında. Bu tanımlanmış DEG’ler, yukarıda tarif edildiği gibi sitokin analizi yapmak için kullanıldı.
MCP kimliği sadece tümör ve bitişik tümör olmayan örnekler kullanarak gerçekleştirdik. TCGA portalından RNA-seq veri kümeleri ve invaziv öncesi akciğer lezyonlarının mikrodizi verileri için MCP’nin genel ekspresyonu hesaplandı45. TCGA veri kümeleri TCGabiolinks kullanılarak indirildi83 R Paket. Sadece ondan fazla tümör veya bitişik tümör olmayan örnekleri olan projeler dahil edildi. İnvaziv öncesi akciğer lezyonlarının veri kümeleri https://github.com/ucl-respiratory/preinvasive.
Araştırma tasarımı hakkında daha fazla bilgi,Doğa portföyü raporlama özeti Bu makaleye bağlı.
 1
Bilim adamları, havadan co₂ yakalayan canlı yapı materyali yaratıyor
81 kez okundu
1
Bilim adamları, havadan co₂ yakalayan canlı yapı materyali yaratıyor
81 kez okundu
 2
Thomas Lovejoy, ‘Biyoçeşitliliğin vaftiz babası’ 80 yaşında ölüyor
49 kez okundu
2
Thomas Lovejoy, ‘Biyoçeşitliliğin vaftiz babası’ 80 yaşında ölüyor
49 kez okundu
 3
Hawaiʻi Abyssal Nodüller ve İlişkili Ekosistemler Expedition
45 kez okundu
3
Hawaiʻi Abyssal Nodüller ve İlişkili Ekosistemler Expedition
45 kez okundu
 4
Volcano Watch-Kīlauea’da bölümler arası gürlemeler
43 kez okundu
4
Volcano Watch-Kīlauea’da bölümler arası gürlemeler
43 kez okundu
 5
Proje Spot Işığı: USGS Bilim Adamları, iklim belirsizliğinin ortasında temel su verilerini tanımlamak için Kenai Yarımadası topluluklarıyla birlikte çalışır
41 kez okundu
5
Proje Spot Işığı: USGS Bilim Adamları, iklim belirsizliğinin ortasında temel su verilerini tanımlamak için Kenai Yarımadası topluluklarıyla birlikte çalışır
41 kez okundu