
Last Updated on Şubat 13, 2026 by EDİTÖR
Kararlı transgenik zebra balığı hatları, 14 saat açık/10 saat kapalı ışık döngüsüne sahip özel bir su sporları tesisinde 28,5 °C’de tutuldu. Casper Tüm deneylerde aşağıdaki genotipe sahip balıklar kullanıldı: mitfa-GÜZELV600E;s.53−/−;ölü−/−. Balıklara Tricaine (MS-222; stok konsantrasyonu 4 g/l) kullanılarak anestezi uygulandı.−1), balık hareketsiz hale gelinceye kadar seyreltildi. Tüm hayvan prosedürleri Memorial Sloan Kettering Kanser Merkezi Kurumsal Hayvan Bakım ve Kullanım Komitesi (protokol no. 12-05-008) tarafından onaylandı.
Oluşturmak hmgb2a Ve hmgb2b İn vivo kullanım için CRISPR kılavuz RNA (gRNA) plazmitleri, her gen için üç gRNA, zebra balığı için optimize edilmiş U6 gRNA destekleyicileri içeren Geçit giriş vektörlerine alt klonlandı. Ortaya çıkan 3x gRNA plazmidi, Gateway LR klonlaması yoluyla birleştirildi. gRNA/Cas9 aktivitesinin in vivo olarak doğrulanması, Alt-R CRISPR-Cas9 sistemi (Entegre DNA Teknolojileri (IDT)) kullanılarak, tek kılavuzlu RNA’ların (sgRNA’lar) ve saflaştırılmış Cas9 proteininin tek hücreli aşamalı zebra balığı embriyolarına enjekte edilmesiyle gerçekleştirildi. 24 saat sonra beş ila on embriyodan genomik DNA izole edildi ve Alt-R Genom Düzenleme Tespit Kiti (IDT) kullanılarak mutasyon tespiti yapıldı.
Zebra balığı gRNA dizileri:
hmgb2a sgRNA1: 5′-GAAAAGTTCACCGAGGTCCC-3′
hmgb2a sgRNA2: 5′-AAGGTGAAGGGCGACAACCC-3′
hmgb2a sgRNA3: 5′-GACAACCCGGCATCTCTAT-3′
hmgb2b sgRNA1: 5′-CAAACCCAAGGGGAAGACGT-3′
hmgb2b sgRNA2: 5′-CTCAAACTTGACCTTGTCGG-3′
hmgb2b sgRNA3: 5′-AGGAGAAGTTGACGGGCACGT-3′
NT sgRNA: 5′-AACCTACGGGCTACGATACG-3′
Tümörler TEAZ aracılığıyla üretildi68. Oluşturmak hmgb2a/hmgb2b nakavt melanomları için, 3 aydan 6 aya kadar yetişkin balıklar rastgele gruplara ayrıldı ve aşağıdaki plazmitler enjekte edildi: miniCoopR–GFP, mitfa:Cas9, Tol2, U6–sgptena, U6–sgptenb ve 394-zU6–3XsgRNA[hmgb2a] ve 394-zU6–3XsgRNA[hmgb2b] veya 394-zU6–3XsgRNA[NT]. Yetişkin balıklara tricain kullanılarak anestezi uygulandı ve sırt yüzgecinin altına 1 ul plazmit karışımı enjekte edildi, hemen elektroporasyona tabi tutuldu ve iyileşmek için tatlı suya taşındı. Tümör büyümesi her 1-2 haftada bir ZEISS Axio Zoom V16 floresan mikroskobu kullanılarak görüntülendi. Erkek ve dişi hayvanlar eşit oranlarda kullanıldı. Herhangi bir örneklem büyüklüğü hesaplaması veya körleme yapılmamıştır.
Aşağıdaki hücre çizgileri Amerikan Tipi Kültür Koleksiyonundan elde edildi: A375 (CRL-1619), SKMEL5 (HTB-70), MIA-PaCa-2 (CRM-CRL-1420), Panc-1 (CRL-1469), HTB-4 (T24), HTB-9 (5637) Ve HEK293T. Hücreler 37 °C ve %5 CO2’de tutuldu2 nemlendirilmiş inkübatör. Hücre dizilerinin kimliği Amerikan Tipi Kültür Koleksiyonu tarafından doğrulandı ve rutin olarak yabancı maddelerden arındırılmış olup olmadığı kontrol edildi. Mikoplazma. Hücreler, %10 fetal sığır serumu (GeminiBio; 100-500) ile desteklenmiş DMEM’de (Gibco; 11965) kültürlendi.
Aşağıdaki genleri hedef alan siRNA’lar Horizon Discovery’den elde edildi: HMGB2 (L-011689-00-0005), SYNE2 (L-019259-01-0005) ve hedeflemesiz kontrol (D-001810-10-05). Durum başına 250.000 A375 hücresini transfekte etmek için DharmaFECT 1 Transfeksiyon Reaktifi (Horizon Discovery; T-2001) kullanıldı. Ortam 24 saat sonra değiştirildi ve deneyler, ortam değiştirildikten 72 saat sonra yapıldı. HMGB2 nakavt, HMGB2’yi hedef alan bir antikorla (MilliporeSigma; HPA053314) western blot ile doğrulandı. Jel kaynağı verileri için Ek Şekil 2’ye bakınız.1. Aşağı regülasyonu SYNE2 aşağıdaki primerlerle kantitatif polimeraz zincir reaksiyonu (qPCR) aracılığıyla doğrulanmıştır:
SYNE2 F: 5′-CAAAGCACAGGAAACTGAGGCAG-3′
SYNE2 R: 5′-AGACAGTGGCAACGAGGACATG-3′
β-Aktin F: 5′-CACCAACTGGGACGACAT-3′
β-Aktin R: 5′-ACAGCCTGGATAGCAACG-3′
LentiCRISPRv2 sistemi69 kararlı insan nakavt hücre çizgileri ve hedefleme gRNA’ları oluşturmak için kullanıldı HMGB2 veya ATAT1 GeCKO2 kütüphanesinden seçildi. Her bir gRNA’yı içeren oligonükleotidler IDT’den elde edildi ve kısıtlama sindirimi yoluyla lentiCRISPRv2 omurgasına klonlandı. BsmB1 ve Quick Ligase (New England Biolabs) ile ligasyon. Bağlanan plazmitler Stbl3 bakterisine (New England Biolabs) dönüştürüldü ve gRNA yerleştirilmesini doğrulamak için dizilendi. Nihai plazmitler, aşağıda açıklandığı gibi lentiviral transdüksiyon kullanılarak stabil A375 ve SK-MEL-5 çizgileri oluşturmak için kullanıldı.
İnsan gRNA dizileri:
HMGB2 sgRNA1: 5′-CTGCACGAAGAAGGCGTACG-3′
HMGB2 sgRNA2: 5′-AAGATCAAAAGTGAACACCC-3′
ATAT1 sgRNA1: 5′-CCAGAAGAACATCTACAGTG-3′
ATAT1 sgRNA2: 5′-CCTCACTGTAGATGTTCTTC-3′
NT sgRNA: 5′-AACCTACGGGCTACGATACG-3′
HMGB2-GFP plazmidini oluşturmak için, bir pENTR omurgasındaki (Horizon Discovery; OHS5898-202621565) insan HMGB2 kodlama dizisi, In-Fusion Klonlama kullanılarak bir C terminali EGFP etiketi ile birleştirildi. Daha sonra HMGB2-GFP eklentisi, LR Clonase II Plus (Thermo Fisher Scientific) kullanılarak Ağ Geçidi klonlaması yoluyla sitomegalovirüs promotörünü (pLX304; Addgene 25890) içeren bir lentiviral ekspresyon vektörüne aktarıldı. Silme yapıları, pENTR omurgasındaki HMGB2open okuma çerçevesini bir şablon olarak kullanarak In-Fusion Klonlama (Takeda Bioscience) yoluyla oluşturuldu ve daha sonra yukarıda açıklandığı gibi Ağ Geçidi klonlaması yoluyla pLX304’e klonlandı. Primerler şunlardı:
HMGB2-ΔA-kutusu F: 5′-CCAACAAGCCTCCCAAAGGTGATAAGAAGGG-3′
HMGB2-ΔA-kutusu R: 5′-TGGGAGGCTTGTTGGGGTCTCTCTTTACC-3′
HMGB2-ΔB-kutusu F: 5′-CCAATGCTGCCAAGGGCAAAAGTGAAGC-3′
HMGB2-ΔB-kutusu R: 5′-CCTTGGCAGCATTGGGGGTCCTTTTTCTTCCC-3′
HMGB2-Δasidik kuyruk F: 5′-CATATCGTGACCCAGCTTTCTTGTACAAAG-3′
HMGB2-Δasidik kuyruk R: 5′-CTGGGTCACGATATGCAGCAATATCCTTTTC-3′
HMGB2OEHMGB2–GFP, HMGB2ISHMGB2arasındaATAT1IS ve FastFUCCI stabil hücre çizgileri, lentiviral transdüksiyon yoluyla üretildi. FastFUCCI raportör plazmidi Addgene’den (86849) elde edildi. HMGB2 – GFP raportör plazmidi, HMGB2 gRNA+Cas9 plazmidleri, ATAT1 gRNA+Cas9 plazmidleri ve hedeflemeyen gRNA+Cas9 plazmitleri yukarıda açıklandığı gibi birleştirildi. HMGB2OE plazmit Horizon Discovery’den (OHS5897-202616132) elde edildi. Koşul başına sekiz milyon HEK293T hücresi, Effectene Transfeksiyon Reaktifi (QIAGEN) kullanılarak 1.200 ng lentiviral vektör, 600 ng PAX2 plazmidi ve 300 ng MD2 plazmidi ile transfekte edildi. Virüs, transfeksiyondan 24 saat sonra başlayarak toplandı. Viral süpernatan, A375 hücrelerine ortam ve 10 µg ml ile 1:1 oranında eklenmeden önce filtrelendi (0.45 µm filtre)−1 polibrenden. Hücreler 72 saat boyunca enfekte edildi, 24 saat boyunca iyileşmelerine izin verildi ve daha sonra blastisidin (5 µg ml-1) kullanılarak seçildi.−1; 7-10 gün) veya puromisin (1 µg ml−1; 3 gün). Bir floresan raportör ifade eden hücre çizgileri için hücreler, FACSAria III veya FACSymphony S6 hücre sıralayıcıları (BD Biosciences) kullanılarak sıralandı. HMGB2 aşırı ekspresyonu ve CRISPR çizgileri için başarılı transdüksiyon, HMGB2’yi hedef alan bir antikorla (MilliporeSigma; HPA053314) western blot yoluyla doğrulandı. ATAT1 içinIS çizgiler, devrilme qPCR aracılığıyla doğrulandı. Jel kaynağı verileri için Ek Şekil 2’ye bakınız.1.
QPCR primer dizileri:
ATAT1 F: 5′-CACAGTCCCACAGGTGAACA-3′
ATAT1 R: 5′-CTCCCTGCTTGGAGTCTTGG-3′
β-Aktin F: 5′-CACCAACTGGGACGACAT-3′
β-Aktin R: 5′-ACAGCCTGGATAGCAACG-3′
A375, HTB-4 ve HTB-9 hücreleri, bir statik hücre sınırlayıcı (4Dcell) kullanılarak 3 um yükseklikte gece boyunca (yaklaşık 16 saat) hapsedilmeye tabi tutuldu. Pankreas duktal adenokarsinomu hücre çizgileri (MIA-PaCa-2 ve Panc-1), daha büyük boyutları nedeniyle 5 µm yükseklikte sınırlandırılmıştır. Hücreler, fibronektin kaplı cam tabanlı 35 mm’lik tabaklarda (FluoroDish) veya cam tabanlı altı oyuklu plakalarda (MatTek) görüntülemeden 6 saat önce kaplandı. Kapatma uygulanmadan önce hücrelerin bağlanmasına izin verildi. Sınırlandırılmış hücreler 37 °C’de ve %5 CO2’de inkübe edildi2 bir gecede. Canlı görüntüleme için, kaplama hücrelerine görüntülemeden 2-3 saat önce boyalar artı 10 μM verapamil eklendi. Canlı görüntüleme için kullanılan boyalar SiR-tubulin (Spirochrome; 100 nM) ve SiR-DNA (Spirochrome; 250 nM) idi. Hapsedilmeden hemen önce farmakolojik inhibitörler eklendi. Kullanılan inhibitörler Taxol (Tocris; 1097), tubasin (Selleck Chemicals; S2239), nocodazol (Tocris; 1228) ve trichostatin A (MilliporeSigma; T8552) idi. Canlı görüntüleme, LSM 880 (ZEISS) konfokal mikroskopta 37 °C’de ve %5 CO2’de gerçekleştirildi.2×63 büyütmede ve 5-10 dakikalık zamansal çözünürlükte, ZEN Black v.2.3 SP1 yazılımı (ZEISS) kullanılarak. İmmünofloresan için hücreler, aşağıda açıklandığı gibi boyama ve görüntülemeye geçmeden önce oda sıcaklığında 15 dakika boyunca %4 paraformaldehit ile sabitlendi.
Hücre proliferasyonunu test etmek için CyQUANT Doğrudan Kırmızı Hücre Proliferasyon Testi (Thermo Fisher Scientific; C35013) kullanıldı. Hücreler, 96 oyuklu plakalara oyuk başına 500 hücre yoğunluğunda kaplandı ve 72 saat boyunca büyümesine izin verildi. Hücre sayısı, hücre kültürü ortamına 1:1 oranında eklenen CyQUANT Direkt Kırmızı Nüklei Asit Boyası ve Arka Plan Bastırıcı kullanılarak ölçüldü ve yoğunluk, bir plaka okuyucu üzerinde 622 nm’de okundu. İstila analizleri için VitroGel Hücre İstila Tahlili Kiti (TheWell Bioscience; IA-VHM01-1P) ve Cultrex Collagen I Hücre İstila Tahlili kiti (Bio-Techne; 3457-096-K) kullanıldı. Hücreler gece boyunca DMEM’de serumsuz bırakıldı, istila tahlili ekinin üst bölmesine kaplandı ve 18 saat boyunca göç etmelerine izin verildi. Hücre sayısı kristal viyole veya kalsein boyama kullanılarak ölçüldü.
Hücreler, cam CC2 kaplı oda slaytları (Thermo Fisher Scientific) veya fibronektin kaplı cam tabanlı tabaklar (FluoroDish) üzerine kaplandı ve yaklaşık 24 saat boyunca bağlanmasına izin verildi. Hücreler 15 dakika boyunca %4 paraformaldehit ile sabitlendi, PBS içerisinde %0,1 Triton ile geçirgenleştirildi ve oda sıcaklığında 1 saat boyunca %10 keçi serumu (Thermo Fisher Scientific) içerisinde bloke edildi. Kullanılan birincil antikorlar tavşan anti-HMGB2 (Abcam; ab124670), tavşan anti-HMGB1 (Abcam; ab18256), tavşan anti-HMGA1 (Abcam; ab129153), fare anti-a-tubulin (MilliporeSigma; CP06), civciv anti-β-tubulin (Novus Biologicals; NB100-1612), fare idi. anti-asetillenmiş tübülin (MilliporeSigma; 6793), tavşan anti-asetillenmiş tübülin (Cell Signaling Technology (CST); 5335), sıçan anti-tirozinli tübülin (MilliporeSigma; MAB1864-I), fare anti-poliglutamillenmiş tübülin (MilliporeSigma; T9822), fare anti-GFP (Abcam; ab1218), tavşan anti-H3Ac (MilliporeSigma; 06-599), fare anti-Annexin V (Santa Cruz Bioteknoloji; sc-74438), tavşan anti-bölünmüş kaspaz-3 (CST; 9661), tavşan anti-bölünmüş PARP (CST; 5625), tavşan anti-YAP (CST; 14074), fare anti-Twist (Abcam; ab50887), tavşan anti-Salyangoz (CST; 3879), tavşan anti-SMAD3 (Abcam; ab40854) ve tavşan anti-SYNE2 (Abcam; ab204308). Tüm birincil antikorlar 1:200’de kullanıldı. Hücreler, gece boyunca 4 °C’de birincil antikorlarla inkübe edildi, PBS’de yıkandı ve uygun floresan etiketli ikincil antikorla (1:250) inkübe edildi. Alexa Fluor 488 konjuge phalloidin (CST; 8878S) kullanıldığında 1:50 oranında eklendi ve Hoechst 1:1.000 oranında eklendi. Hücreler VECTASHIELD’e (Vektör Laboratuvarları) monte edildi ve gece boyunca kürlenmeye bırakıldı. Lekeli hücreler, ZEN Black v.2.3 SP1 yazılımı (ZEISS) kullanılarak bir ZEISS LSM 880 konfokal üzerinde x40 veya x63 çözünürlükte görüntülendi. Yalnızca görselleştirme amacıyla gürültüyü azaltmak için ara sıra görüntülere 0,5-0,75 piksel yarıçaplı bir Gauss bulanıklığı uygulandı.
İnsan melanom dokusu mikrodizileri TissueArray.Com’dan (Me481f) elde edildi. Slaytlar 60°C’de 20 dakika süreyle pişirildi ve ardışık ksilen ve etanol yıkamalarıyla parafinleri giderildi. Antijen geri kazanımı, düdüklü tencerede 95°C’de 20 dakika ısıtılan 1X IHC Antijen Geri Kazanım Solüsyonu (Thermo Fisher Scientific; 00-4955-58) kullanılarak gerçekleştirildi. PBS’de yıkandıktan sonra numuneler, aşağıdaki birincil antikorlarda gece boyunca 4 °C’de inkübasyondan önce oda sıcaklığında 1 saat boyunca %10 keçi serumunda (Thermo Fisher Scientific) bloke edildi; tümü 1:200’de bloklama tamponunda seyreltildi: tavşan anti-HMGB2 (Abcam; ab124670) ve fare anti-asetillenmiş tübülin (MilliporeSigma; 6793). PBS’de yıkandıktan sonra slaytlar, uygun floresan etiketli ikincil antikor (1:250) ve Hoechst (1:1.000) ile inkübe edildi. PBS’de yıkandıktan sonra, slaytları VECTASHIELD’e yerleştirmeden önce tümör hücrelerini etiketlemek için floresanla konjuge edilmiş bir tavşan anti-S100a6 antikoru (Abcam; ab204028; 1:250) ile son bir inkübasyon gerçekleştirildi. Slaytlar, yukarıda açıklandığı gibi bir LSM 880 konfokal (ZEISS) üzerinde elde edilen daha yüksek çözünürlüklü görüntülerle, ×20/0,8 sayısal açıklık hedefi kullanılarak bir Pannoramik slayt tarayıcı (3DHISTECH) üzerinde görüntülendi.
Görüntüler CellProfiler kullanılarak analiz edildi70TrackMate71 ve MATLAB v.R2021b ve R2023b (MathWorks). Sabit hücrelerin görüntüleri için hücreler, bir çekirdek maskesi oluşturmak için Hoechst boyaması ve bir tam hücre maskesi oluşturmak için falloidin veya başka hücre iskeleti boyaması kullanılarak CellProfiler’da bölümlere ayrıldı. Hücre/çekirdek başına ortalama yoğunluk ölçüldü ve nükleer lokalize proteinlerin ekspresyonu, çekirdek başına Hoechst yoğunluğuna göre normalleştirildi. Canlı görüntüleme verilerinin miktarının belirlenmesi için zaman içinde hücre başına HMGB2-GFP yoğunluğu TrackMate kullanılarak ölçüldü. Ortaya çıkan yoğunluk verileri, her eğriye bir çizgi yerleştirilerek ve dörtten fazla veri noktasının en iyi uyum çizgisinden 0,2 au’dan fazla farklı olduğu eğrileri otomatik olarak kaldırarak MATLAB’da analiz edildi. Tüm durumlarda çizim ve istatistikler MATLAB’da yapıldı. Görüntüler Fiji (v.2.14) kullanılarak şekil hazırlığı için bir araya getirildi.
HMGB2-GFP’yi eksprese eden A375 hücreleri, FRAP ölçümlerinden önce yaklaşık 18 saat süreyle sınırlandırıldı. FRAP, LSM 880 konfokal üzerinde 37 °C’de %5 CO2 ile yapıldı2 ×63 yağlı daldırma lens ve ZEN Black v.2.3 SP1 yazılımı (ZEISS) kullanılarak. On darbe için 405 nm ve 488 nm dalga boylarında foto-ağartmadan önce her hücrenin çekirdeği içinde 5 µm’lik dairesel çaplı bir ilgi alanı tanımlandı. Işıkla ağartmadan önce bir zaman noktası elde edildi. Floresans geri kazanımı, toplam 20 saniye boyunca 0,2 saniyelik aralıklarla görüntülendi. Tüm analizler MATLAB’da yapıldı. Analiz için, ilgilenilen bölgedeki floresans, başlangıç zaman noktasındaki (foto-ağartmadan önce) floresansa normalleştirildi. İlgili bölgedeki floresansın ön ağartma değerinin en az %25’ine kadar ağartılmamış olduğu numuneler otomatik olarak analizden çıkarıldı. Her bir kurtarma eğrisi, ‘exp2’ parametresi ile ‘fit’ fonksiyonu kullanılarak iki bileşenli bir üstel ile donatıldı: F(T) =sen0 +A1(1 –e−T/ T1) + A2(1 – e− T/ T2), Neresi sen0, ışıkla ağartma işleminden hemen sonraki floresansı temsil eder, A1 hızla yayılan popülasyonun genliğini temsil eder,A2 yavaş yayılan popülasyonun genliğini temsil eder, T zamanı geldi ve T1 ve T2 sırasıyla hızlı yayılan ve yavaş yayılan popülasyonların zaman sabitlerine karşılık gelir.
Hücreler, cam tabanlı Petri kaplarına (FluoroDish FD35) yerleştirildi ve yukarıda açıklandığı gibi 18 saat süreyle hapsedildi. Tabağın sınırlayıcıdan çıkarılmasından hemen sonra hücre sertliği, QI Gelişmiş modunda bir NanoWizard V mikroskobu (JPK Bruker) kullanılarak ölçüldü. PetriDishHeater (Bruker) kullanılarak görüntüleme sırasında numuneler 37 °C’de tutuldu. Hücre sertliği haritalaması için 1 μm çapında küresel AFM probu (silikon nitrür konsol; nominal yay sabiti) k= 0,2 Nm−1; SAA-SPH-1UM; Bruker) kullanıldı. AFM probunun her bir yay sabiti, 37 °C’de sıvı içinde termal gürültü yöntemi kullanılarak ölçülmüştür. Sertlik haritalaması için, cam yüzey etkisini önlemek amacıyla %10-20’ye kadar numune girintisi sağlamak amacıyla 2 nN’lik bir ayar noktası kullanıldı (32 x 32 piksel çözünürlüklü 60 μm x 60 μm görüntü boyutu). Veriler, uyum parametresi olarak 0,5 Poisson oranına sahip Hertz modeli kullanılarak JPK Veri İşleme yazılımı ile işlendi. Nükleer sertliği hesaplamak için, nükleer bölgeyi çıkarmak üzere karşılık gelen hücre yüksekliği ölçümleri temel alınarak kuvvet haritaları bölümlere ayrıldı.
TurboID yapılarının klonlanması ve doğrulaması, önceki bir çalışmada açıklandığı gibi gerçekleştirildi.35. Sito-TurboID plazmidi Addgene’den elde edildi. TurboID-HMGB2 için, TurboID kaseti polimeraz zincir reaksiyonuyla güçlendirildi ve In-Fusion Klonlama kullanılarak HMGB2’nin N terminalinde pENTR-HMGB2’ye klonlandı. Nükleer lokalizasyon sinyali-TurboID için, TurboID kasetini içeren In-Fusion Klonlama kullanılarak bir giriş vektörü oluşturuldu ve bunu ardışık üç nükleer lokalizasyon sinyal dizisi takip etti. pENTR – TurboID – HMGB2 ve pENTR – TurboID – 3XNLS yapıları, LR Clonase II Plus (Thermo Fisher Scientific) kullanılarak Ağ Geçidi klonlaması yoluyla pLX304 omurgasına alt klonlandı. Yukarıda açıklandığı gibi stabil A375 hücre çizgileri oluşturuldu ve TurboID füzyon proteininin ekspresyonu ve lokalizasyonu, hemaglutinin’i (TurboID kasetinin N terminalinde bulunur) hedef alan immünofloresan ile doğrulandı. TurboID aktivitesi, hücrelere 10 mM biyotin (MilliporeSigma; 1071508) darbesi uygulanarak ve ardından floresan etiketli streptavidin kullanılarak hem western blotlama hem de immünofloresan kullanılarak doğrulandı. Western blotlama için IRDye 800CW Streptavidin (LI-COR; 926-32230) kullanıldı ve immünofloresan için Alexa Fluor 488 (Thermo Fisher Scientific; S11223) veya Alexa Fluor 555’e (Thermo Fisher Scientific; S21381) konjuge edilmiş streptavidin kullanıldı.
Kütle spektrometresi deneyleri için, koşul ve kopya başına on milyon hücre, 15 cm’lik tabaklara kaplandı. Ortam tabaklardan çıkarıldı ve 1 saat boyunca 10 mM biyotin ile değiştirildi. Etiketleme reaksiyonu, tabakların buz üzerine yerleştirilmesi ve hücrelerin buz soğukluğunda PBS ile beş kez yıkanması yoluyla durduruldu. Hücreler daha sonra buz soğukluğunda PBS içerisinde kazınarak ayrıldı ve daha sonra topaklandı ve radyo-immünopresipitasyon tahlil tamponu + proteaz inhibitörleri içerisinde yeniden süspanse edildi. Hücreler sonikasyon (%10 amplitüd; döngü başına 2 saniye; altı döngü) aracılığıyla lize edildi ve protein konsantrasyonunu ölçmek için Bradford tahlili kullanıldı. Her numune için 1 mg protein, radyo-immünopresipitasyon tahlil tamponunda streptavidin manyetik boncuklarla (Thermo Fisher Scientific; 88817) gece boyunca 4 °C’de rotasyonla inkübe edildi. Ertesi gün, boncuklar manyetik bir raf kullanılarak topaklandı, süpernatan çıkarıldı ve boncuklar bir kez 50 mM Tris-HCl (pH 7,5) içinde ve iki kez 50 mM Tris-HCl (pH 7,5) içindeki 2 M üre içinde yıkandı.
Boncuklar 80 ml 2 M üre ve 50 mM EPPS (pH 8,5) içerisinde yeniden süspanse edildi ve dl-ditiyotreitol (1 mM son konsantrasyon), bir Thermomixer (Thermo Fisher Scientific) üzerinde çalkalanarak (1.100 rpm) 37 °C’de 30 dakika süreyle. Serbest sistein kalıntıları, karanlıkta 1.100 rpm’de 25 °C’de 45 dakika boyunca 2-iyodoasetamid (3.67 mM nihai konsantrasyon) ile alkile edildi. Reaksiyon, 3.67 mM ditiyotreitol kullanılarak söndürüldü ve LysC (750 ng) ilave edildi, ardından 1.150 rpm’de 37 °C’de 1 saat süreyle inkübasyon yapıldı. Son olarak, trypsin (750 ng) ilave edildi ve ardından 1.150 rpm’de 37 °C’de 16 saat süreyle inkübasyon yapıldı. İnkübasyondan sonra sindirim, %50 trifloroasetik asit (TFA) ilavesiyle pH 3’ün altına kadar asitleştirildi ve peptitler, Sep-Pak C18 kartuşları (Waters) üzerinde tuzdan arındırıldı. Kısaca, kartuşlar sırasıyla (1) %100 metanol; (2) %70 asetonitril (ACN)/%0,1 TFA; ve (3) iki kez %5 ACN/%0,1 TFA. Şartlandırmanın ardından asitleştirilmiş peptid sindirimi kartuşa yüklendi. Sabit faz iki kez %5 ACN/%0.1 formik asit ile yıkandı. Son olarak peptitler, %70 ACN/%0,1 formik asit kullanılarak iki kez elüe edildi. Elüsyona tabi tutulan peptitler, bir SpeedVac santrifüjünde vakum altında kurutuldu, ardından 12 ul %0,1 formik asit içerisinde sulandırıldı, sonikasyon yapıldı ve bir otomatik numune alıcı şişesine aktarıldı. Peptit verimi NanoDrop (Thermo Fisher Scientific) kullanılarak ölçüldü.
Peptitler, C18 sabit fazından (IonOpticks; Aurora 3 1801220) oluşan, 75 mm çapında ve 1,7 mm parçacık boyutuna sahip 25 cm’lik bir sütun üzerinde, 90 dakika boyunca %2 ila %35 B’ye ve ardından 7 dakika boyunca %95 B’ye (tampon A, yüksek performanslı sıvı kromatografi dereceli suda %0,1 formik asit; tampon B, tampon B, %99,9 ACN ve %0,1 formik asit) 300 nl dk akış hızıyla−1 nanoElute 2 sistemi (Bruker) kullanılarak. Kütle spektrometresi verileri, CaptiveSpray kaynağı (Bruker) ile bir timsTOF HT (Bruker) üzerinde, veriden bağımsız bir satın alma paralel birikim-seri parçalanma (PASEF) yöntemi (dia-PASEF) kullanılarak elde edildi. Kütle aralığı 100’den 1700’e ayarlandı M/zve iyon hareketlilik aralığı 0,60 V s cm2’ye ayarlandı−2 (çarpışma enerjisi 20 eV) ila 1,6 V s cm−2 (çarpışma enerjisi 59 eV) 100 ms rampa süresi ve 100 ms birikim süresi ile. Dia-PASEF ayarları, 400,0–1.201,0 Da’lık bir kütle aralığını, 0,60–1,60’lık hareketlilik aralığını ve 1,80 saniyelik bir döngü süresi tahminini içeriyordu. Dia-PASEF pencereleri, 26.00 Da’lık bir kütle genişliğine, 1.00 Da’lık bir kütle örtüşmesine ve döngü başına 32 kütle adımına ayarlandı.
Ham veri dosyaları Spectronaut v.18.5 (Biognosys) kullanılarak işlendi ve 15 Ağustos 2023’te (226.261 giriş) indirilen bir insan UniProt protein veritabanıyla Pulsar arama motoruyla arandı. Sistein karbamidometilasyonu sabit bir modifikasyon olarak belirtilirken metiyonin oksidasyonu, protein N terminalinin asetilasyonu ve deamidasyon (NQ) değişken modifikasyonlar olarak belirlendi. Maksimum iki trypsin kaçırılan bölünmeye izin verildi. Aramalarda peptit FDR’yi kontrol etmek için ters dizi tuzak stratejisi kullanıldı ve tanımlama için eşik olarak %1 FDR belirlendi. EşleştirilmediT -test hesaplamak için kullanıldı P diferansiyel analizdeki değer ve log bazında volkan grafiği oluşturuldu2FC ve Q değer (birden fazla test düzeltildi P Benjamini-Hochberg yöntemini kullanarak değer). A Q ≤ 0,05 değeri istatistiksel olarak anlamlı kesme noktası olarak kabul edildi.
Fare in vivo çalışmaları, Memorial Sloan Kettering Kanser Merkezi Kurumsal Hayvan Bakım ve Kullanım Komitesi ve Araştırma Hayvan Kaynakları Merkezi tarafından onaylanan kurallara uygun olarak gerçekleştirildi. Fareler patojen içermeyen koşullar altında, sıcaklığın (21,5 °C ± 1,5 °C) ve nemin (%55 ± %10) kontrollü olduğu bir ortamda ve 12 saatlik aydınlık/karanlık döngüsü altında barındırıldı. İlaç etkililik çalışmaları için, 6 ila 8 haftalık atimik dişi farelere (Jackson Laboratuvarı), Matrigel (Corning) ile 50:50’lik bir karışım halinde beş milyon A375 hücresi deri altından enjekte edildi. Tümörler ortalama 100 mm hacme ulaştığında3fareler iki tedavi grubuna randomize edildi ( N= grup başına 4-6 fare) araç kontrolü veya trametinib (1 mg kg-1) almak üzere−1) dabrafenib (30 mg kg-1) ile kombinasyon halinde−1). Her iki ilaç da 3 hafta boyunca günde beş kez oral sondayla verildi. Fareler, hastalık/ölüm belirtileri açısından tedavi süresi boyunca günlük olarak gözlemlendi. Tümörler kumpas kullanılarak haftada iki kez ölçüldü ve hacim, aşağıdaki formül kullanılarak hesaplandı: uzunluk x genişlik2× 0,52. Vücut ağırlığı da haftada iki kez değerlendirildi. HMGB2 silme yapısı büyüme eğrisi çalışmaları için, yukarıda açıklandığı gibi tümör hücreleri implante edildi ve tümör hacmi haftada iki kez ölçüldü. Hayvanlar, tümör boyutları 1.500 mm’ye ulaşana kadar izlendi.3Bu noktada tümörler toplandı, 24 saat boyunca %10 formalinde sabitlendi, %70 etanole aktarıldı ve histoloji için işlendi. Memorial Sloan Kettering Kanser Merkezi (MSKCC) Kurumsal Hayvan Bakım ve Kullanım Komitesi tarafından belirlenen sınırlara uygun olarak, tümör yükü 1.500 mm3’ü aştığında farelere ötenazi uygulandı.3 . Hiçbir deneyde bu sınırlar aşılmadı. Histoloji, immünohistokimya için aşağıdaki antikorlar kullanılarak HistoWiz tarafından gerçekleştirildi: fare anti-BRAFV600E(Abcam; ab228461) ve tavşan anti-asetillenmiş tübülini (Abcam; ab179484). Her iki deney için de koşul başına dört ila altı fareden (grup başına toplam 10-11 fare) oluşan iki biyolojik kopya gerçekleştirildi. Herhangi bir örneklem büyüklüğü hesaplaması veya körleme yapılmamıştır.
HMGB2’yi aşırı eksprese eden A375 ve SKMEL5 hücrelerinin toplu RNA sekansı için, her biri yaklaşık bir milyon hücreden oluşan üç kopya topaklandı ve hızlı dondurmadan önce TRIzol içinde yeniden süspanse edildi. Sınırlandırılmış A375 hücrelerinin toplu RNA sekansı için, altı oyuklu bir plakanın her bir oyuğuna 200.000 hücre kaplandı. Üç kuyucuk, 3 µm yükseklikte altı kuyulu statik sınırlayıcı (4Dcell) kullanılarak yaklaşık 18 saat süreyle sınırlandırıldı, geri kalan üç kuyu ise sınırlandırılmadan bırakıldı. Hücreler daha sonra TRIzol’de toplandı ve her biri yaklaşık 600.000 hücreden oluşan numuneler oluşturmak üzere her durum için üç kuyucuk bir araya getirildi. Bu işlem, koşul başına toplam üç bağımsız biyolojik kopya için tekrarlandı. Kütüphane hazırlığı ve sıralaması Azenta Life Sciences tarafından yapıldı. Ham sıralama okumaları FastQC (Babraham Bioinformatics) ve Trimmomatic kullanılarak işlendi72 insan genomu hg38’e hizalanmadan önce. Tüm aşağı akış analizleri R (v.4.3.1)’de gerçekleştirildi. Diferansiyel gen ekspresyonu DESeq2 (ref.73) varsayılan parametrelerle. GSEA fgsea kullanılarak yapıldı74 MSigDB’den Gene Ontology biyolojik süreç yolu setlerini içeren R paketi (v.1.26)75.
Her biri yaklaşık 100.000 hücre içeren numuneler 700°C’de santrifüj edildi. G %10 DMSO ile desteklenmiş 500 ul büyüme ortamında yeniden süspanse edilmeden önce 4°C’de 5 dakika süreyle. Hücreler, kütüphane hazırlığı ve sıralama Azenta Life Sciences tarafından gerçekleştirilmeden önce gece boyunca -80 °C’de donduruldu. Sıralama okumaları, kalite kontrolü için 15, Cutadapt kalite ayarıyla TrimGalore (v.0.6.7) kullanılarak kırpıldı ve filtrelendi76 (v.4.0) ve FastQC v.0.12.1. Okumalar, Bowtie 2 (ref. 2) kullanılarak insan genom düzeneği hg38 ile hizalandı.77) (v.2.3.5.1) ve Picard’dan (Broad Institute; v.2.16) MarkDuplicates kullanılarak tekilleştirildi. Zirveler MACS2 kullanılarak tanımlandı (ref.78) ile P-kamuya açık bir melanosit veri kümesi kullanılarak 0,001’lik değer ayarı (GSM3191792) kontrol olarak. Küresel bir zirve atlası oluşturmak için, 500 bp’lik bir bölgedeki tüm zirveler birleştirilmeden ve featureCounts kullanılarak okumaların niceliği belirlenmeden önce kara listeye alınan bölgeler kaldırıldı. Farklı olarak zenginleştirilmiş tepe noktaları DESeq2 (ref.73). Tepe gen haritalaması, tüm genler arası tepe noktalarının o gene atanmasıyla ve diğer durumlarda, transkripsiyon başlangıç bölgesine genomik mesafeyle yapıldı. Yol, ClusterProfiler kullanılarak analiz edildi79. Kasırga grafikleri deepTools ile oluşturuldu80 (v.3.5.1) işlevleri (computeMatrix ve graphHeatmap), belirtilen yol kümelerinden açıklamalı genlerle birlikte. Motif zenginleşmesi Homer kullanılarak analiz edildi81 (v.4.11.1) işlevleri (findMotifsGenome ve annotatePeaks).
A375 ve SKMEL5 hücrelerinde HMGB2 bağlanmasının profilini oluşturmak için, yeni toplanan hücreler (yaklaşık 20 milyon hücre/kopyalama/durum), önce oda sıcaklığında 20 dakika boyunca 1,5 mM EGS (Thermo Fisher Scientific; 21565) ile ve ardından 4 °C’de 40 dakika boyunca %1 formaldehit (Thermo Fisher Scientific; 28906) ile çapraz bağlandı. Reaksiyon, 0,125 M nihai konsantrasyona kadar glisin ilave edilerek söndürüldü. Sabit hücreler, iki kez PBS ile yıkandı ve SDS tamponunda (100 mM NaCl, 50 mM Tris-HCl (pH 8,0), 5 mM EDTA, %0,5 SDS ve 1x proteaz inhibitör kokteyli; Roche) yeniden süspanse edildi. Ortaya çıkan çekirdekler döndürüldü, immünopresipitasyon tamponunda (100 mM NaCl, 100 mM Tris-HCl (pH 8.0), 5 mM EDTA ve %5 Triton X-100) 2:1 oranında karıştırılmış 0,5 milyon hücre başına 1 ml’de yeniden süspanse edildi, 1 x proteaz inhibitör kokteyli (MilliporeSigma; 11836170001). Çekirdekler, aşağıdaki parametrelerle ortalama 200-300 bp parça uzunluğu elde etmek için bir Covaris E220 Odaklanmış ultrasonik cihazda işlendi: tepe olay gücü = 140, görev faktörü = 5, patlama başına döngü/saniyede patlama = 200 ve zaman = 20 dakika (A375 hücreleri için) veya 45 dakika (SKMEL5 hücreleri için). Kromatin konsantrasyonları, üreticinin talimatlarına göre Pierce BCA Protein Test Kiti (Thermo Fisher Scientific; 23227) kullanılarak tahmin edildi. İmmünopresipitasyon reaksiyonları, Protein LoBind Tüplerinde (Eppendorf; 22431081) 500 ul immünopresipitasyon tamponunda kuruldu ve 4 °C’de 2 saat boyunca 50 ul Dynabeads Protein G (Thermo Fisher Scientific; 10004D) ile önceden temizlendi. Ön temizlemenin ardından numuneler yeni Protein LoBind Tüplerine aktarıldı ve gece boyunca 4 °C’de 5 µg HMGB2 (Abcam; ab67282), V5 (Abcam; ab9116; yalnızca SKMEL5 ChIP için kullanıldı) ve H3K4me3 (Epicypher; 13-0041) antikorlarıyla inkübe edildi. Normalleştirme amacıyla 5 µlDrosophila Her reaksiyona spike-in kromatin (Active Motif; 53083) ve 2 ul spike-in antikoru (Active Motif; 61686) ilave edildi. Ertesi gün, her reaksiyona 50 ul BSA ile bloke edilmiş Dynabeads Protein G ilave edildi ve 2 saat boyunca 4 °C’de inkübe edildi. Boncuklar daha sonra iki kez düşük tuzlu yıkama tamponu (150 mM NaCl, %1 Triton X-100, %0,1 SDS, 2 mM EDTA ve 20 mM Tris-HCl (pH 8,0)) ile, iki kez yüksek tuzlu yıkama tamponu (500 mM NaCl, %1 Triton X-100, %0,1 SDS, 2 mM EDTA ve 20 mM) ile yıkandı. Tris-HCl (pH 8,0), iki kez LiCL yıkama tamponu (250 mM LiCl, 10 mM Tris-HCl (pH 8,0), 1 mM EDTA, %1 Na deoksikolat ve %1 IGEPAL CA-630) ve bir kez TE tamponu (10 mM Tris-HCl (pH 8,0) ve 1 mM EDTA) ile. Numuneler daha sonra elüsyon tamponunda (%1 SDS ve 0.1 M NaHCO3) gece boyunca ters çapraz bağlandı.3) ve üreticinin talimatları takip edilerek ChIP DNA Clean & Concentrator kiti (Zymo Research; D5205) kullanılarak saflaştırıldı. Geri kazanılan DNA fragmanlarının miktarının belirlenmesinden sonra, üreticinin talimatlarını takip ederek ThruPLEX DNA-Seq Kiti (Takara Bio; R400676) kullanılarak kütüphaneler hazırlandı, SPRIselect manyetik boncuklarla (Beckman Coulter; B23318) saflaştırıldı ve bir Qubit Flex florometre (Thermo Fisher Scientific) kullanılarak miktarı belirlendi ve TapeStation (Agilent) kullanılarak profili oluşturuldu. Kütüphaneler, Illumina NovaSeq 6000’de (kitaplık başına yaklaşık 30-40 milyon 100 bp eşleştirilmiş uç okuma) sekanslama yapılması için MSKCC Integrated Genomics Operation çekirdek tesisine gönderildi.
ChIP-seq okumaları, kalite ayarı ve 15 kalite ayarıyla TrimGalore (v.0.4.5) kullanılarak ve Cutadapt çalıştırılarak kalite ve kitaplık bağdaştırıcıları için kırpıldı ve filtrelendi.76 (v.1.15) ve FastQC (v.0.11.5). Okumalar, Papyon 2 (v.2.3.4.1) (ref.77) ve Picard Tools’daki (v.2.16.0) MarkDuplicates kullanılarak tekilleştirildi. Zenginleştirilmiş bölgeleri belirlemek için MACS2 (ref.78) ile kullanıldıP -değer ayarı 0,001’dir ve her koşul için eşleşen bir kontrole karşı çalıştırılır. BEDTools paketindeki (v.2.29.2) ‘birleştirme’ işlevi kullanılarak tüm tepe noktalarının üst kümesinin birleştirilmesiyle bir tepe atlası oluşturuldu. Okuma yoğunluğu profilleri, deepTools ‘bamCoverage’ (v.3.3.0) kullanılarak oluşturuldu, on milyon benzersiz şekilde eşlenmiş okumaya normalleştirildi ve okuma yığınları 200 bp’ye çıkarıldı. Ham sayım matrisi oluşturmak için featureCounts (v.1.6.1) aracı kullanıldı ve kopyalarla yapılan deneyler için tüm ikili kontrastlar için diferansiyel zenginleşmeyi hesaplamak üzere DESeq2 kullanıldı. Tek örnekli veriler için MACS2, diferansiyel bölgeleri bulmak amacıyla farklı koşullardaki bam’lar değiştirilerek çalıştırıldı. Tepe gen ilişkileri, tüm intragenik tepe noktalarının o gene atanmasıyla oluşturulurken, intergenik tepe noktaları, transkripsiyon başlangıç bölgesine olan doğrusal genomik mesafe kullanılarak atanmıştır. GSEA82 her gene en büyük (büyüklük olarak) loga sahip tek zirvenin atandığı, önceden sıralanmış seçenek ve varsayılan parametrelerle gerçekleştirildi.2FC bununla bağlantılı. DeepTools kullanılarak kompozit ve kasırga grafikleri oluşturuldu80 (v.3.3.0), 25 bp’lik pencerelerde örneklenen ortalama sinyal ve çevreleyen 2 kb tarafından tanımlanan yan bölge ile normalleştirilmiş kodamanlar üzerinde computeMatrix veplotHeatmap’i çalıştırarak. Homer kullanılarak motif imzaları elde edildi81 (v.4.5).
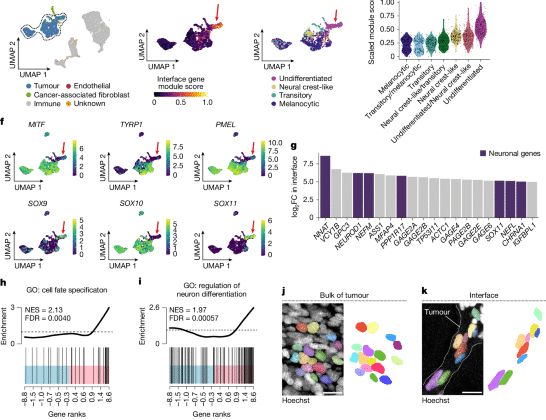
Önceki bir çalışmadan insan melanomu scRNA-seq verileri7 Gene Expression Omnibus’tan (GEO) indirildi (GSE115978). Tüm analizler Seurat kullanılarak R’de yapıldı.83 (v.4.4.0 ve v.5.0.1). Sayım matrisi sctransform kullanılarak normalleştirildi. Kümeleme, 0,8 çözünürlükte Seurat işlevleri (FindNeighbors ve FindClusters) kullanılarak yapıldı. Hücre türleri ve tedavi durumu, orijinal yayındaki meta veriler kullanılarak açıklandı7. Hücre tipleri önceki bir çalışmadaki gen listeleri kullanılarak sınıflandırıldı2 ve varsayılan parametrelerle Seurat işlevi AddModuleScore. Modül puanları 0 ile 1 arasında ölçeklendirildi. Hücreler, verilen gen modülleri için en yüksek ifade puanı esas alınarak farklılaşma durumuna göre sınıflandırıldı. Diferansiyel olarak eksprese edilen genler, varsayılan parametrelerle Seurat fonksiyonu FindMarkers kullanılarak hesaplandı. GSEA, yukarıda açıklandığı gibi fgsea kullanılarak gerçekleştirildi.
Tüm istatistiksel analizler ve çizim, R (RNA-seq ve ATAC-seq verileri için; v.4.3.1) veya MATLAB’da (görüntüleme verileri için; v.R2021b) gerçekleştirildi. ScRNA-seq verileri için,P değerler, Wilcoxon sıralama toplamı testi ve çoklu gruplar için Bonferroni düzeltmesi (R fonksiyonu çift yönlü.wilcox.test) kullanılarak hesaplandı. Pearson korelasyon katsayıları ve karşılık gelenP değerler R fonksiyonu kor.testi kullanılarak hesaplandı. Toplu RNA-seq ve toplu ATAC-seq verilerinin diferansiyel ekspresyon analizleri için,Pdeğerler Wald testi kullanılarak DESeq2’de hesaplandı. Görüntü analizi için,Pdeğerler Tukey post hoc testi kullanılarak MATLAB fonksiyonları (anova1 ve multicompare) kullanılarak hesaplandı. Hücre göç hızını hesaplamak için, tek tek hücrelerin zaman noktaları boyunca kat ettiği Öklid mesafesi, MATLAB fonksiyonu pdist kullanılarak ölçüldü ve hız, zaman adımına bölünerek hesaplandı. Fare deneylerinde, büyüme hızı farklılıklarını araştırmak amacıyla bir dizi olabilirlik oranı testi gerçekleştirdik. Araç verilerine tek bir üstel uyum ile erken ve geç zaman büyüme oranlarına ilişkin tahminler elde etmek için maksimum olabilirlik tahmini kullanılarak büyüme eğrisine iki üstel bir model oturtuldu. Gösterilen tüm temsili görüntüler için görüntüler en az üç bağımsız kopyayı temsil eder.
Araştırma tasarımına ilişkin daha fazla bilgi şu adreste mevcuttur:Doğa Portföyü Raporlama Özeti bu makaleye bağlı.
 1
HEMOROİD – DR. DEVRİM DEMİREL VE DR. GAFFAR KARADOĞAN
1189 kez okundu
1
HEMOROİD – DR. DEVRİM DEMİREL VE DR. GAFFAR KARADOĞAN
1189 kez okundu
 2
Jurnal ve Jurnalcilik ne demek? Osmanlı’dan günümüze ihbarcılık
195 kez okundu
2
Jurnal ve Jurnalcilik ne demek? Osmanlı’dan günümüze ihbarcılık
195 kez okundu
 3
Hindistan’ı yöneten cesur Türk kadını: Raziye Sultan’ın tarihi hikayesi
177 kez okundu
3
Hindistan’ı yöneten cesur Türk kadını: Raziye Sultan’ın tarihi hikayesi
177 kez okundu
 4
Avustralya ‘Çöp Papağanları’ artık yerel bir ‘içme geleneği’ geliştirdi
161 kez okundu
4
Avustralya ‘Çöp Papağanları’ artık yerel bir ‘içme geleneği’ geliştirdi
161 kez okundu
 5
Papa Francis’in son vasiyeti ortaya çıktı: Yeni Papa Leo’nun ilk rotası Türkiye mi olacak?
158 kez okundu
5
Papa Francis’in son vasiyeti ortaya çıktı: Yeni Papa Leo’nun ilk rotası Türkiye mi olacak?
158 kez okundu