
Aşağıdaki kimyasallar ve reaktifler kullanıldı: Dounce homojenleştirici (DWK Life Sciences, 885302-0002); Pierce anti-ha manyetik boncuklar (Thermo Scientific, 88837); Pierce anti-flag manyetik agaroz (Thermo Scientific, A36797); anti-Flag M2 manyetik boncuklar (Sigma Millipore, M8823); Pierce Protein A/G manyetik boncukları (Thermo Scientific, 88802); İGepal CA-630 (Sigma-Aldrich, I8896); S-Trap mikro sütunlar (Protifi, C02-mikro-80); trietilamonyum bikarbonat (TeAB) tamponu (Sigma-Aldrich, T7408); sodyum dodesil sülfat (SDS; Bio-Rad, 1610302); DSSO (Thermo Scientific, A33545); DHSO (CF Plus Chemicals, PCL042); DMTMM (Sigma-Aldrich, 74104); N-Dodesil β-D-Maltosid (DDM, Altın Biyoteknolojiler, DDM5); NativeMark Protein Standardı (Invitrogen, LC0725); Ana sayfa% 4-6 jeller (Invitrogen, BN1002box); Çok ekranlı filtre plakaları (Sigma Millipore, MSHVN4510); TMTPRO 16PLEX Seti (Thermo Fisher Scientific, A44520); proteaz inhibitörü kokteyli (Roche, 4906845001); Tris (2-karboksietil) fosfin (TCEP; altın biyoteknoloji, 51805-45-9); 2-kloroasetamid (Sigma-Aldrich, C0267); S-Metil tiyometanesülfonat (MMTS; Sigma-Aldrich, 208795); tripsin (Promega, v511c); Lys-C (Wako Chemicals, 129-02541); hidroksilamin çözeltisi (Sigma-Aldrich, 438227); SEP-PAK C18 ve C8 50 mg kartuş (Waters, WAT054955 ve WAT054965); yüksek PH ters fazlı peptit fraksiyonlama kiti (Thermo Scientific, 84868); Bio-Rad protein test boyası (Bio-Rad, 5000006); 3-[4-(2-hydroxyethyl)-1-piperazine]propanesülfonik asit (Thermo Scientific, J61296AE); C18 (Sigma Millipore, 66883-U); Gateway LR Clonaz II enzim karışımı (Thermo Scientific, 11791020); NebNext Ultra II Q5 Master Mix (New England Biolabs, M0544L); CAS9-NLS (Kaliforniya Üniversitesi, Berkeley’de QB3 Macrolab); Kloner (Stemcell Technologies, 05889); MISEQ Reaktif Nano Kit V2 (300 döngü; Illumina, MS-103-1001); Geneart Precision Grna Sentez Kiti (Thermo Fisher Scientific, A29377); Rnaeasy Qiagen kiti (Qiagen, 74104); 24 oyuklu cam tabanlı plakalar (Cellvis, p24-1.5hn); Corning Square Kültür Yolu (Corning, 431110); Nunc Nunclon Delta Hücre Kültürü Yemekleri (Thermo Scientific, 140675, 150318 ve 168381); Corning Matrigel Matrisi (Corning, 354230); F-12 ile DMEM (Gibco, 11330057); nörobasal ortam (Thermo Scientific, 21103049); esassız amino asitler (Gibco, 11140050); Glutamax (Gibco, 35050061); N-2 takviyesi (Gibco, 17502048); nörotrofin-3 (NT3; Peprotech, 450-03); beyin kaynaklı nörotrofik faktör (BDNF; Peprotech, 450-02); B27 (Gibco, 17504001); Y27632 dihidroklorür (kaya inhibitörü; Peprotech, 1293823); Cultrex 3D Kültür Matrisi Laminin I (Ar-Ge Systems, 3446-005-01); Accutaz (Stemcell Technologies, 07922); FGF2-G3 (şirket içi); insan insülin (Santa Cruz Biotechnologies, SC-360248); Dönüştürücü Büyüme Faktörü-y (Peprotech, 100-21); holo-transferrin insanı (Sigma-Aldrich, T0665); sodyum bikarbonat (Sigma-Aldrich, S5761-500G); sodyum selenit (Sigma-Aldrich, S5261-10G); Doksisiklin (Clontech Labs, 631311); Ultraür 0.5 M EDTA (Invitrogen, 15575020); % 16 paraformaldehit (elektron mikroskopi bilimi, 15710); DMEM (Gibco, 11995073); fetal sığır serumu (Cytiva, SH30910.03); hidrokortizon (Sigma-Aldrich, H0135); polietilenimin (Polissisler, 23966); Fugene (Promega, E2311).
Aşağıdaki birincil antikorlar kullanıldı (immünoblotlama için 1: 1,000, immünofloresan için 1: 400): Flag (Sigma-Aldrich, F1804), HA (Hücre Sinyali Teknolojisi, 3724), 3724), 3796-82), D5, TMEM230 (origenen, TA50488-82), D2, D230 (origenten, TAM1), DiRicen (Hücre Sinyali Teknolojisi, C8B1), CLR (ProteIntech, 10292-1-AP), Golgin 97 (ProteIntech, 12640-1-AP), VDAC1 (ProteIntech, 55259-1-AP), Clcn3 (hücre sinyal teknolojisi, 133599s), MCH (termo bilimsel), MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MCH, MSH, (Hücre sinyal teknolojisi, C45B10). Aşağıdaki ikincil antikorlar kullanıldı (immünoblotlama için 1: 10.000, immünofloresan için 1: 400): anti-tavşan immünoglobulin-G (IgG) at turp peroksidaz (HRP) konjugatı (Bio-Rad, 1706515); anti-fare IgG HRP konjugatı (Bio-Rad, 1706516); keçi anti-chicken Igy (H + L), Alexa Fluor 488 (Thermo Scientific, A-11039); Keçi anti-rat IgG (H + L) Çapraz adsorbed, Alexa Fluor 555 (Thermo Scientific, A-21434); Keçi anti-tavşan IgG (H + L) Çapraz Adsorbed, Alexa Fluor 647 (Thermo Scientific, A-21244).
Plazmidler daha önce tarif edildiği gibi yapıldı61. Human Orfeome Collection, sürüm 8’den gelen giriş klonları, Gateway Technology (Thermo Fisher Scientific) veya Gibson Assembly (New England Biolabs) kullanılarak karşılık gelen plazmidlerine klonlandı. Tam TMEM230 (Y29C/R78L/X121W) mutant, gen sentezi (Twist Bioscience) ile elde edildi. Lentivirüs transdüksiyonu için faj ve plenti omurgaları kullanıldı. Transfeksiyon için PCG’ler ve PCDNA3.1 omurgaları kullanıldı. Aşağıdaki plazmidler üretildi: PGCS-3 × Flag-ATP11b (Addgene, 225511), PCDNA-TMEM30A-V5 (Addgene, 225510), PGCS-3 × HA-TMEM230 (AddGene, 22551230 (Y29/R71), PGCS-3 x HA-TMEM230 (PGCS-3 × 225513), Plenti-UBC-HA-TMEM230 (AddGene, 225516), Plenti-UBC-HA-TMEM230 (R78L) (Addgene, 225517), Plenti-UBC-HA-TMEM230 (X121W) 225520), Plenti-OBC-HA-TMEM230 (X121PG) (Addgene, 225518), Plenti-UBC-HA-TMEM230 (Addgene 225521), PCDNA-Clcn3-3 × Bayrak (AddGen, 225506), PCD-CLC, PCDA-CLC, PCD-CLC, PCD55-3 × Bayrak (AddGen, 225506), 225507), PCDNA-TMEM9B-3 × HA (AddGene, 225509), PCDNA-TMEM9-3 × HA (Addgene, 225508), faj-MCH-CLCN3 (Addgene, 225514), phage-TMEM9-EGFP (AddGene, 22515). Lentiviral ambalaj için aşağıdaki plazmidler kullanıldı: PPAX2 (Addgene, 12259), PMD2 (Addgene, 12260).
HEK293 hücreleri (ATCC; RRID: CVCL_0045),% 10 fetal sığır serumu ile desteklenmiş yüksek glikoz ve piruvat DMEM ile 10 cm’lik yemeklerde kültürlendi. CO-IP deneyleri için hücreler, polietilenimin (25 kDa) kullanılarak 2: 1 oranında 6 ug plazmid ile% 60 konfluipte transfekte edildi ve 48 saat 37 ° C’de ve% 5 CO’da inkübe edildi2. Sum159pt hücreler (T. Walter’dan bir hediye, Memorial Sloan Kettering; RRID: CVCL_5423), DMEM’de 6 oyuklu kültür yemeklerinde (oyuk başına 300.000 hücre), glutamax ile takviye edilmiş F-12 ile% 5 fetal sığır serumu, 1 μg−1 hidrokortizon ve 5 ug ml−1 insülin. Hücreler 1 gün sonra Fugen ve Optimem Transfeksiyon reaktifi kullanılarak 500 ng plazmid ile transfekte edildi ve 37 ° C ve% 5 CO’da inkübe edildi2. Transfeksiyondan bir gün sonra, hücreler puromisin ile seçildi ve 24 oyuklu cam tabanlı kültür yemeklerine (oyuk başına 50.000-100.000 hücre) kaplandı.
Gen tarafından düzenlenen insan embriyonik kök (ES) hücreleri (H9, Wicell Enstitüsü) daha önce tarif edildiği gibi kültürlendi62–63. Hücreler, Matrigel ile kaplanmış plakalar üzerinde E8 ortamı ile tutuldu ve DPBS içinde 0.5 mM EDTA ile bölündü. ATCC, kısa tandem tekrar analizi kullanarak HEK293T hücre hattının kimlik doğrulamasını sağlamak için kalite testi gerçekleştirir. H9 ES hücreleri (Wicell’den), G-band karyotipleme ve kısa tandem tekrar analizi kullanılarak Wicell tarafından doğrulanır. Genetik olarak düzenlenen H9 insan ES hücreleri karyotipleme ile doğrulandı. HEK293, SUM159T ve H9 hücre dizileri, mikoplazma için Mycoplasma artı PCR test kiti (Agilent 302107) kullanılarak aylık olarak test edildi. Bu çalışma için H9 hücrelerinin kullanımı Embriyonik Kök Hücre Araştırma Gözetim Komitesi (onay numarası 00051) tarafından onaylanmıştır.
AAVS1-TRE3G-NGN2 sürücüsü ile insan ES hücreleri64 daha önce tarif edildiği gibi eleneuronlara farklılaştırıldı65. Kısaca, kök hücreler 2 x 10’da kaplandı5 Hücreler ML−1 (Farklılaşma Günü 0) ND1 ortamında (F-12, N-2, İnsan 10 ng ml ile DMEM−1 Bdnf, 10 ml−1 İnsan NT3, esassız olmayan amino asitler, 0.2 ug ml−1 2 mg ml ile desteklenmiş insan laminin)−1 Doksisiklin ve 10 uM Y27632 (kaya inhibitörü). Ertesi gün, ortam Y27632 olmadan ND1 ile değiştirildi. Ertesi gün, ortam ND2 (nörobasal ortam, b27, glutamax, 10 ng ml ile değiştirildi−1 Bdnf, 10 ml−1 Nt3) 2 ug ml ile desteklenmiştir−1 Doksisiklin. Deney gününe (19-21 gün) kadar, ortamın% 50’si her gün taze ND2 ile değiştirildi. Hücreler 4 x 10’da tekrarlandı5 4. günlerde kuyu başına hücreler. 10. günden itibaren doksisiklin ND2’den çıkarıldı.
Lentiviral vektörler, daha önce tarif edildiği gibi HEK293T hücrelerinde (ATCC numarası CRL-3216; RRID: CVCL_0045) paketlendi62–66–67. Hücreler, polietilenimin kullanılarak 4: 2: 1 oranında PPAX2, PMD2 ve hedef vektör ile% 60 konfluansta birlikte transfekte edildi. Ortam ertesi gün ND2 olarak değiştirildi ve transfeksiyondan 2 gün sonra toplandı. Lentivirüs içeren ND2 ortamı süzüldü (0.22 uM) ve farklılaşma gün 11-12’de ineuronların transdüksiyonu için kullanıldı.
İnsan ES hücreleri (H9 AAVS1-TRE3G-NGN2 3 × FLAG-EEA1; RRID: CVCL_D1KV) CRISPR-CAS9 (Ref.68). Hücreler, daha önce tarif edildiği gibi bir neon transfeksiyon sistemi kullanılarak 0.6 μg kılavuz RNA ve 3 μg Cas9-NL (QB3 Macrolab, California Üniversitesi, Berkeley) karışımı ile elektroporasyona uğradı.69 Ref.70. Homozigot insan ES hücreleri üretmek için TMEM230X121w variant, a single-stranded DNA oligonucleotide was included in the electroporation (5′-CTACCGTGGTTACTCCTATGATGACATTCCAGACTTTGATGACTGGCACCCACCCCATAGCTGAGGAGGAGTCACAGTGGAACTGTCCCAGCTTTAAGATATCTAGCAGAAACTATAGCTG-3′). Hücreler, düşük bir oda 24-48 saat boyunca geri kazanıldı2 İnkübatör ve Sony Biyoteknoloji (SH800S) hücre sıralayıcısı (RRID: SCR_018066) ile tek hücrelere ayrılmıştır. Tek tek klonların gen düzenlenmesi, Illumina Miseq sistemi (RRID: SCR_016379) ile sekanslama ile doğrulandı ve immünoblotlama ve/veya MS ile valide edildi. Kılavuz RNA’lar diziler için Geneart Precision GrNA sentez kiti (Thermo Fisher Scientific) kullanılarak oluşturuldu: TMEM230-/ – 5′-CCTGAAGGTCAATGTAGCCATCGT-3 ′, TMEM230X121w 5′-CTCCTCCTCAGCTATGGGGT-3 ′, TMEM9-/ – 5′-TATCTTTGGTGGCTGTGGTC-3 ′, TMEM9B-/ – 5′-TCTACATCAGGCCCCCGCAC-3 ′. Burada bildirilen ES hücreleri talep üzerine sunulacak, ancak Wicell’den bir malzeme transfer anlaşması gerektirecektir.
İmmünofloresan boyama için Sum159pt hücreler, PBS içinde% 4 paraformaldehid ile 15 dakika sabitlendi ve oda sıcaklığında 10 dakika boyunca PBS içinde% 0.5 Triton X-100 ile geçirgenleştirildi. Hücreler, oda sıcaklığında 1 saat boyunca% 0.1 Triton X-100 ile PBS içinde% 3 BSA ile bloke edildi. Hücreler, 4 ° C’de 3 saat boyunca% 0.1 Triton X-100 ile PBS içinde% 3 BSA içerisinde primer antikorlar (1: 200 seyreltme) ile inkübe edildi. Yıkamalardan sonra hücreler, 4 ° C’de 1 saat boyunca Alexa Fluor sekonder antikorları (1: 400) ile inkübe edildi ve çekirdekler, Hoechst 33342 (1: 10.000) ile 5 dakika boyandı. Hücreler yıkandı ve mikroskopi analizine kadar PBS içinde tutuldu. Bilelerin immüno -lekelenmesi, Ref.71.
Hücreler, bir Nikon Eclipse Ti-E motorlu mikroskop ve bir Plan Apochromat 100 × 1.45 NA yağ-objektif lens üzerinde bir Yokogawa CSU-X1 eğirme-disk konfokal kullanılarak görüntülendi. Canlı hücre görüntüleme, 37 ° C,% 5 CO’da bir Tokai Hit Stage Top İnkübatör ile gerçekleştirildi2 ve% 95 nem. Görüntüler bir Hamamatsu Orca-Fusion BT CMOS kamera ile elde edildi (6.5 μm2 Fotodiot, 16-bit) ve NIS-Elements görüntü edinme yazılımı (RRID: SCR_002776). Tüm numuneler aynı pozlama süresi ve lazer gücü altında ölçüldü. Ortak lokalizasyon analizi, ImageJ/Fiji (RRID: SCR_002285) için JACOP eklentisi (RRID: SCR_025164) ile yapıldı.72 Maksimum yoğunluklu projeksiyon görüntüleri ve maksimum entropi eşiği kullanma. Aynı kültürden elde edilen görüntüleri ve aynı biyolojik kopyadan elde edilen görüntüleri hesaba katmak için LME4 R paketinde yuvalanmış bir tasarıma sahip doğrusal karışık etki modeli istatistikleri uygulanmıştır. Üç bağımsız biyolojik kopyanın her biri için görüş alanlarının sayısı rakamlarda gösterilmiştir (Şek. 4e, f).
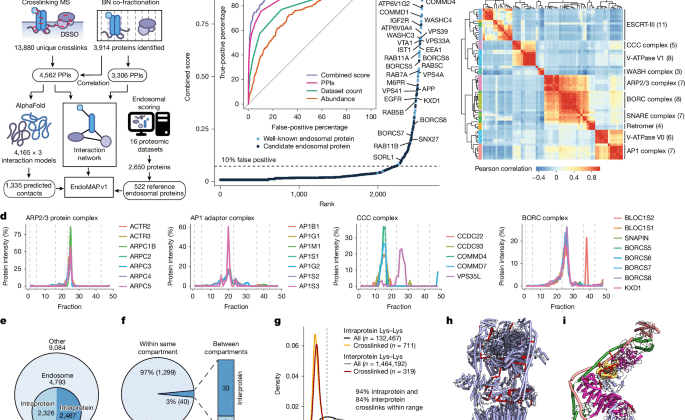
Skorlama yöntemi, endozomal proteomu tanımlamak ve her proteine, deneysel verilere dayanarak endozomlarda bulunma olasılığını yansıtan tarafsız bir skor atamak için gerçekleştirildi. Literatür, memeli organizmalarında endozomal proteomu yakalayan çalışmalar için incelendi, bu da 16 veri kümesiyle sonuçlandı18–19–73–74–75–76–77–78–79–80–81–82 (Ek Tablo 1). Eksik veri kümeleri veya belirsiz organel arıtmaları (örneğin, ‘veziküller’ veya karışık organeller) hariç tutulmuştur. Eski uniprot kimlikleri ve eski gen adları güncellendi (UNIPROT 2022-02). Ensembl ve BioMart R paketi (RRID: SCR_019214), kemirgen genlerini çoklu gen eşleştiğinde tüm insan genleri de dahil olmak üzere insan ortologlarına almak ve eşleştirmek için kullanıldı. Sonraki analizler, puanlama yöntemi için bir metrik olarak veri kümeleri arasındaki protein tanımlamasına dayanıyordu (Ek Tablo 1). Puanlama metriklerinin ve veri kümelerinin performansını değerlendirmek için, 292 iyi bilinen endosomal proteinin referans listesi, yayınlanmış literatürden manuel olarak küratörlüğünü yaptı.1–3–22–56–83–84–85–86–87–88–89–90–91–92–93–94–95–96–97–98–99–100–101–102–103–104–105–106–107–108–109–110–111–112–113–114–115–116 (Genişletilmiş veriler Şekil 1A). Veri kümesine genel bakış, Factominer R paketi (RRID: SCR_014602; Genişletilmiş Veri Şekil. 1b). Çeşitli organeller konumlarına protein ek açıklaması, önceki bir çalışmaya dayanıyordu62 (Genişletilmiş Veri Şekil. 1C). Endozomal skorlamanın bir başka metriği, aşağıda tarif edildiği gibi endozomal peletlerin etiketsiz proteomik analizinden elde edilen endo-IP’deki protein bolluğu idi (Genişletilmiş Veri Şekil. 1d ve ek masa 1). Endozomal proteinlerle etkileşim sayısı Bioplex 3.0 (RRID: SCR_016144), Stringdb ve Corum’dan (28.11.2022 Corum 4.1 sürüm) elde edildi.28–117–118 Yukarıda tarif edilen iyi bilinen endozomal proteinlerin referans listesi için. StringDB için, sadece deneysel kanıtlarla veya yüksek puanla veritabanlarına (birleşik skor> 0.7) fiziksel etkileşimler dahil edildi.
Endozomal proteinleri (yukarıda tarif edilen referans listesinden) sınıflandırmak için her bir metriğin performansı, prediktör olarak binom lojistik regresyonu olan Proc R paketi kullanılarak alıcı çalışma karakteristik eğrileri ile değerlendirildi (Şek. 1b). Kombine endozomal skor, üç ayrı metriğin toplamı ile elde edildi. Eğrinin altındaki kısmi alan ve bir proteini endozomal olarak değerlendirme eşiği, referans listesine dayanarak% 10 yanlış pozitifti. Skorlama yöntemi, toplam 522 protein için iyi bilinen endosomal proteinin referans listesi ile birleştirilen 407 öngörülen endozomal protein (Mitocarta3.0’da bulunan 14 protein (RRID: SCR_018165) hariç tutuldu) ile sonuçlandı. 1). Bu proteinler Bioplex 3.0 kullanılarak karakterize edildi (Ref.28), OpenCell (istiyorum: SCR_021870)119ve UniProt’tan alınan yayınlar (Genişletilmiş Veri Şekil. 1e -g). Sonraki tüm analizler için endozomal ek açıklama bu listeye dayanmaktadır.
HEK293 ile Endo IPSO Hücreler daha önce tarif edildiği gibi yapıldı120. Hek293O FLAG – EEA1 eksprese eden hücreler (Ref.19) birlikte fraksiyonlama deneyleri için kopya başına beş 24.5 cm kare kültür yemekinden toplanmıştır (N = 3) ve çapraz bağlama deneyleri için kopya başına 60 kare plaka (iki partiye ayrılmıştır) (N = 2). Bilelerdeki endo IPS daha önce tarif edildiği gibi yapıldı44–121. Dereuronlardaki deneyler için kopya başına üç 15 cm kültür yemeği kullanıldı (N = 3). Hücreler 1.000’de peletlendiG 4 ° C’de 2 dakika boyunca ve bir kez KPBS tamponu (100 mM potasyum fosfat, 25 mM KCl ve proteaz inhibitörü kokteyl, pH 7.2) ile yıkandı. Hücre peletleri KPBS içinde yeniden süspanse edildi ve 25 vuruşlu bir Dounce homojenleştirici içinde lize edildi. Numuneler 1.000’de iki kez santrifüjlendiG 4 ° C’de 5 dakika boyunca ve PNS protein konsantrasyonu ölçüldü ve Bradford deneyi ile normalleştirildi. Numuneler, 4 ° C’de 50 dakika süreyle 70 uL anti-Flag Sigma manyetik boncukları, 1.6 mL Sigma anti-Flag Sigma manyetik boncukları ve ortak fraksiyonlama deneyleri için 20 mL anti-Flag pierce manyetik boncuklar için parti başına parti başına parti başına inkübe edildi. Boncuklar, KPBS ile manyetik bir stand kullanılarak dört kez yıkandı. Kantitatif proteomik için endozomlar, 4 ° C’de 30 dakika boyunca KBPS içinde 120 uL% 0.5 NP40 (IGEPAL) ile elüte edildi ve MS numune hazırlanmasına kadar -80 ° C’de saklandı. Birlikte fraksiyonlama ve çapraz bağlama deneyleri için endozomlar, 45 dakika boyunca 45 dakika boyunca KPBS içinde 0.8 mM 3 x Flag peptidi ile iki kez elüte edildi (Genişletilmiş Veri Şekil. 1 saat). Peptit ile elili numuneler, 10.000’de 20 dakika santrifüjlendiGPosi-tıknaz tüplerinde (Denville, C2170). Endozomal peletler, fazla 3 × bayrak peptidini uzaklaştırmak için iki kez KPBS ile yıkandı ve hemen işlendi. MS analizindeki kapsamı artırmaya yardımcı olan çapraz bağlama deneyinin ikinci kopyası için ek bir yıkama yapıldı.
Bu analiz için bir protokol ref.122. HEK293 hücrelerinin 10 cm’lik bir kültür tabağından veya kopya başına 15 cm’lik bir kılıftan proteinler (N= 2 veya 4), 25 mM HEPES pH 7.4, 150 mM NaCl ve proteaz inhibitörü kokteyl içinde% 0.5 DDM ile 4 ° C’de 1 saat ekstrakte edildi.123. Örnekler 20.000’de iki kez santrifüjlendiGve süpernatan, 4 ° C’de 2 saat boyunca protein etiketine bağlı olarak 15 uL anti-HA manyetik boncuklar (Pierce) veya 25 uL anti-Flag manyetik boncukları (Sigma) ile inkübe edildi. Endojen antikorlar kullanılarak IP için süpernatan, 15 uL manyetik A/G boncukları ile inkübasyondan önce gece boyunca 5 ug antikor ile inkübe edildi. Boncuklar manyetik bir stand ile ayrıldı ve dört kez yıkama tamponu ile yıkandı (% 0.1 DDM, 25 mM Hepes, 150 mM NaCl, pH 7.4). Boncuklara bağlı proteinler, immünoblotlama için 30 uL 1.5 × Laemmli tamponu veya MS analizi için 30 μl 1.5 x S-tuzlu lizis tamponu (% 7.5 SDS, 150 mM TeAB pH 8.5) ile elüte edildi ve 5 dakika boyunca 80 ° C’de ısıtıldı.
Laemmli tamponu ile karıştırılmış numuneler, 80 ° C’de 5 dakika süreyle inkübe edildi ve sonraki immünoblotlama için bir kriter TGX lekesiz prekast jel içine yüklendi. Elektroforezden sonra, jeller bir Bio-Rad Chemidoc görüntüleyici (Bio-Rad) kullanılarak tarandı ve bir gece boyunca bir PVDF membranına elektro-transfer edildi. Membranlar% 5 yağsız süt ile bloke edildi ve 4 ° C’de 2 saat boyunca primer antikor ile inkübe edildi ve daha sonra HRP-conjugated antikorları ile 4 ° C’de hrp-conjugated antikorlar ile inkübe edildi. Yıkamadan sonra, SuperSignal West Pico Plus Chemilüminesans substratı (Thermo Fisher, katalog numarası 34580) kullanılarak bir biyo-rad Chemidoc görüntüleyicide leke görüntüleri elde edildi. Görüntüler Bio-Rad Image Lab yazılımı ile işlendi (sürüm 6.1.0; RRID: SCR_014210). Yüklemedeki farklılıklar, toplam protein miktarının lekesiz nicelendirilmesi kullanılarak normalleştirildi. Bu prosedür için protokoller ref.124. Tüm jel ve lekelerin tam versiyonları Ek Şekil. 1.
Bu prosedür için ayrıntılı bir protokol Ref.125. Üç bağımsız biyolojik endo-IP kopyasından elde edilen protein kompleksleri, daha önce tarif edildiği gibi kesildi ve işlendi126. Taze hazırlanmış saflaştırılmış endozomal topaklar,% 0.5 DDM ile 40 ul KPBS içerisinde yeniden süspanse edildi ve proteinler, 45 dakika boyunca 4 ° C’de rotasyonda ekstrakte edildi. Protein özleri, 20.000’de santrifüj ile netleştirildiGve 10 uL BN yükleme tamponu, 1 uL Coomassie G-250 karışımı ve 0.5 uL doğal moleküler ağırlık markeri ile karıştırılır. Numuneler, 1.5 saat boyunca 150 V’da% 4-16 ana sayfa jel ve 4 ° C’de 20 dakika boyunca 250 V’da çalıştırıldı. Jeller% 50 etanol ve% 3 fosforik asit içinde sabitlendi, ardından Coomassie ile boyandı. Her numune 48 1 mm dilim halinde kesildi ve jel içi sindirim için 96 oyuklu bir filtre plakasına aktarıldı123. Kısaca, proteinler, 50 mM amonyum bikarbonatta 30 dakika boyunca 37 ° C’de 100 uL 5 mM TCEP ile azaltıldı. Proteinler, oda sıcaklığında 15 dakika boyunca 50 mM amonyum bikarbonat içerisinde 20 mM kloroasetamid ile alkillendi. Fraksiyonlar, 37 ° C’de 4 saat boyunca 0.2 ug Lys-C ile kararlaştırıldı, kurutuldu ve sindirildi, ardından 0.2 ug tripsin ile gece boyunca inkübasyon yapıldı. Peptitler ekstrakte edildi, bir SpeedVac içinde kurutuldu ve veri bağımsız edinme (DIA) sıvı kromatografisi (LC) –M/MS analizi için% 5 asetronitril (ACN),% 5 formik asit içerisinde yeniden oluşturuldu.
Her iki çapraz bağlama prosedürü için ayrıntılı bir protokol Ref.127. İki bağımsız biyolojik replikattan taze hazırlanmış saflaştırılmış endozomal pelet, 300 ul KPBS içinde yeniden süspanse edildi ve hemen 1 mM DSSO (disuccinimidil sülfoksit (2,5-dioksopopopirrolidin-1-il) ile inkübe edilerek hemen çapraz bağlandı. ester) -sulfoksit, Thermo Fisher Scientific) oda sıcaklığında 40 dakika boyunca (ref.6). Reaksiyon, 30 dakika boyunca oda sıcaklığında 50 mM Tris tamponu pH 7.5 ile söndürüldü. Çapraz bağlı numuneler, 8 m üre içinde denatüre edildi, 37 ° C’de 30 dakika boyunca 5 mM ditiotreitol ile azaltıldı ve oda sıcaklığında 30 dakika boyunca 40 mM kloroasetamid ile alkillendi. Çapraz bağlı proteinler, gece boyunca 37 ° C’de Lys-C (1:75) ile sindirildi. Numune üre konsantrasyonu, 50 mm 3- ile 2 m’ye seyreltildi.[4-(2-hydroxyethyl)-1-piperazine]propanesülfonik asit ve 37 ° C’de tripsin (1: 100) ile 6 saat inkübe edildi. Peptitler, 50 mg C8 SEP-PAK katı faz ekstraksiyon kolonu ile tuzdan arındırıldı, kurutuldu ve güçlü katyon değişim kromatografisi ile fraksiyone edildi. Mobil fazın 70 dakikalık doğrusal bir gradyanı (% 20 ACN’de 0.5 M NaCl,% 0.05 formik asit) 14 dakikada% 0 ila 8, 28 dakikada% 20, 48 dakikada% 40’a ve 0.18 mL dakikalık bir kolon akış hızında 68 dakikada% 90’a kullanıldı.−1 Polylc polisülfoetil bir kolonda (3 uM parçacık boyutu, 2.1 mM iç çap ve 100 mm uzunluk). Fraksiyonlar her 30 saniyede her 30 saniye başlayarak 10 dakika ve daha sonra her dakika toplandı. Fraksiyonlar bir SpeedVac içinde kurutuldu ve bir C8 stagretip kullanılarak tuzdan arındırıldı. Her numune için yaklaşık 30 fraksiyon% 5 ACN,% 5 formik asit içinde yeniden oluşturuldu ve LC -MS/MS ile analiz edildi.
Taze hazırlanmış saflaştırılmış endozomal peletin ek bir bağımsız biyolojik kopyası, 300 ul KPBS içinde yeniden süspanse edildi ve 90 dakika boyunca 37 ° C’de 8 mM DHSO ve 16 mM DMTMM kombinasyonu ile inkübe edilerek hemen çapraz bağlandı (ref.32). Çapraz bağlı numuneler% 5 SDS içinde denatüre edildi ve kısaca sonike edildi, 55 ° C’de 5 dakika boyunca 5 mM ditiotreitol ile azaltıldı ve 20 mM mmt ile alkillendi. Çapraz bağlı proteinler çökeltildi ve üretici tarafından sağlandığı gibi S-Trap mini-spin kolon sindirim protokolüne tabi tutuldu (aşağıya bakınız). Peptitler tuzdan arındırıldı ve yukarıda tarif edildiği gibi güçlü katyon değişim kromatografisi ile fraksiyone edildi. Toplam 30 fraksiyon LC -MS/MS ile analiz edildi.
PNS numunelerinin üç bağımsız kopyası (deneye bağlı olarak 10 ug veya 50 ug protein) ve endo-IP numuneleri eşit hacimde su ile karıştırıldı ve numune hazırlamaya tabi tutuldu. S-Trap Micro-Spin kolon sindirim protokolü (sürüm 4.7), üretici tarafından sağlandığı gibi takip edildi (Protifi, C02-Micro-80)128–129–130. Kısaca, her numune eşit hacimlerde 2 x lizis tamponu (% 10 SDS, 100 mm TeAB tamponu pH 8.5) ile karıştırıldı. Ineuronlardan protein IP örnekleri (N= 2 veya 4) doğrudan 1.5 × liziz tamponu içinde toplandı. Proteinler, 55 ° C’de 30 dakika süreyle 5 mM TCEP ile inkübe edilerek azaltıldı ve 40 mM kloroasetamid ile oda sıcaklığında 30 dakika alkillendi. Numuneler fosforik asit ile asitleştirildi ve yıkama tamponu (% 90 metanol, 100 mM TeAB tamponu pH 7.55) ile karıştırıldı. Numuneler mikro spin kolonlarına aktarıldı ve santrifüj ile 150 ul yıkama tamponu ile 4 kez yıkandı. Proteinler, nemli bir odada gece boyunca 37 ° C’de 0.5 ug Lys-C ile sindirildi, ardından 0.5 ug tripsin ile 6 saat inkübasyon. Peptitler kolondan sonraki üç santrifüj aşaması (50 mM TeAB tamponu, sırasıyla% 0.2 formik asit ve% 50 ACN ile) ile toplandı ve bir speedVAC’de kurutuldu.
Peptitlerin etiketlenmesi için protokoller ref.131. Peptitler 50 uL (PNS numuneleri) veya 35 uL (endo-IP numuneleri) 100 mm TeAB tamponu pH 8.5 içerisinde yeniden süspanse edildi. PN’ler ve endo-IP peptitleri, 11 uL veya 7 ul ACN ilave edilerek ve 10 uL veya 8 uL TMTPRO reaktifi (20 mg mL ile oda sıcaklığında 1 saat inkübe edildi.−1ACN’de stok). Reaksiyon, 15 dakika boyunca% 10 uL% 5 hidroksilamin ilave edilerek söndürüldü.
PNS numuneleri için, her numune için eşit peptit miktarları birleştirildi, 100 mg C18 SEP-PAK katı faz ekstraksiyon kolonu ile tuzdan arındırıldı ve temel pH ters faz-performanslı LC ile fraksiyone edildi. Kromatografi, 10 mM amonyum bikarbonat pH 8’de% 5 ila% 35 ACN arasında 50 dakikalık doğrusal bir gradyan ile gerçekleştirildi.−1 Agilent 300 kullanılarak C18 kolonunu genişletin (3.5 uM parçacık boyutu, 2.1 mm iç çap ve 250 mm uzunluk). Toplanan ilk 96 fraksiyon, daha önce tarif edildiği gibi 24 fraksiyon halinde birleştirildi132. Bir setin bitişik olmayan fraksiyonu bir speedVac içinde kurutuldu ve C18 stagretip kullanılarak tuzdan arındırıldı. Kurutulmuş peptitler% 5 ACN,% 5 formik asit içinde yeniden oluşturuldu ve LC -MS/MS analizine tabi tutuldu.
Endo-IP numuneleri için, her numune için eşit peptit miktarları birleştirildi ve üreticinin protokolünü takiben yüksek PH ters fazlı peptit fraksiyonlama kiti (Pierce) kullanılarak fraksiyone edildi. Eluatlar, C18 Stagretip kullanılarak kurutuldu ve tuzdan arındırıldı. Kurutulmuş peptitler% 5 ACN,% 5 formik asit içinde yeniden oluşturuldu ve LC -MS/MS analizine tabi tutuldu.
Protein IP numuneleri için, her numune için eşit peptit miktarları birleştirildi, kurutuldu ve daha fazla fraksiyonlama olmadan C18 stagretip kullanılarak tuzdan arındırıldı. Kurutulmuş peptitler% 5 ACN,% 5 formik asit içinde yeniden oluşturuldu ve LC -MS/MS analizine tabi tutuldu.
TMT etiketli numuneler, Faims Pro (Ref.131). Peptitler, 20 cm ACCUTORE C18 reçinesi (2.6 μm, 150 A) ile paketlenmiş 100 uM mikrokapiller kolon üzerinde ayrıldı. 80 dakikada% 5 ila% 20 ACN, 83 dakikada% 36 ve% 0.125 formik asitte 85 dakikada% 98’e 90 dakikalık doğrusal bir gradyan kullanıldı.−1 . MS1Spectra orbitrap’ta edinildi (60.000 çözünürlük, tarama aralığı 350-1.350M/zıplastandart otomatik kazanç kontrolü (AGC) hedefi, otomatik maksimum enjeksiyon süresi). Peptit fragmantasyonu,% 36 normalleştirilmiş çarpışma enerjisinde yüksek enerjili çarpışma ayrışması (HCD) ile elde edildi. MS2 Spektrumlar orbitrap’ta edinildi (30.000 çözünürlük, izolasyon penceresi 0.6M /zıplaTurbotmt Tüm TMT reaktiflerine ayarlanmış, ilk kütle 120M /zıpla,% 200 normalize edilmiş AGC, 120 ms maksimum enjeksiyon süresi). Faims Pro, −30, −50 ve −70 telafi voltajı (CV) olarak ayarlandı. Fraksiyone edilmemiş numuneler (protein IP’ler), ikinci çalışma için −40, −60 ve −80 CV’ye ayarlanmış Faims ile iki kez enjekte edildi.
BN-PAGE birlikte fraksiyonlama numuneleri, bir Orbitrap Exploris 480 kütle spektrometresine (RRID: SCR_022215) bağlı bir kolay-NLC 1200 sistemi kullanılarak analiz edildi. 15 cm 100 μm kılcal kolon, Accucoore 150 C18 reçinesi (2.6 μm, 150 Å) ile şirket içinde paketlendi. 80 dakikada% 5 ila% 20 ACN, 83 dakikada% 25’e ve% 0.125 formik asitte 85 dakikada% 98’e 90 dakikalık doğrusal bir gradyan kullanıldı.−1 . DIA yöntemi MS’den oluşuyordu224’ün üst üste binen izolasyon pencerelerinin analiziM /zıpla390–1,014’ten geçtiM /zıplaİlk döngü için kütle aralığı ve 402-1,026M/zıplaİkinci döngü için133. DIA taramaları% 28 normalleştirilmiş HCD çarpışma enerjisi, 30.000 çözünürlük, 145-1.450 ile yapıldı M/zıplaTarama aralığı,% 1.000 normalleştirilmiş AGC ve 54 ms maksimum enjeksiyon süresi. Bunu bir ebeveyn MS izledi1 iyon taraması (60.000 çözünürlük, tarama aralığı 350-1,050 M/ zıpla,% 100 normalleştirilmiş AGC hedefi, otomatik maksimum enjeksiyon süresi).
DSSO-Crosslinking numuneleri, Faims Pro (RRID: SCR_020562) ile bir Orbitrap Fusion Lumos kütle spektrometresine bağlanmış bir kolay-NLC 1200 sistemi kullanılarak analiz edildi. 80 dakikada% 5 ila% 20 ACN, 83 dakikada% 25, 85 dakikada% 40’a ve% 0.125 formik asitte 2 dakika boyunca% 98’e 90 dakikalık doğrusal bir gradyan 0.5 uL dakikada kullanıldı−1 . Bir HCD-MS2 stratejisi kullanıldı4MS’in1Spektrum orbitrap’ta elde edildi (120.000 çözünürlük, tarama aralığı 400-1.600M/ zıplastandart AGC hedefi, otomatik maksimum enjeksiyon süresi). Şarj durumları 4-8 olan peptitler,% 21, 27 ve% 33 normalleştirilmiş çarpışma enerjisinde HCD ile parçalandı. MS2 orbitrap’ta edinildi (60.000 çözünürlük, izolasyon penceresi 1.6M/zıplaOtomatik tarama aralığı,% 200 normalleştirilmiş AGC, 120 ms maksimum enjeksiyon süresi). Faims Pro −50, −60 ve −75 CV olarak ayarlandı (Ref.134).
DHSO- ve DMTMM geçişli örnekler, Faims Pro ile Orbitrap Ascend ACCEND Multiomics Tribid kütle spektrometresine bağlı bir Vanquish Neo UHPLC sistemi kullanılarak analiz edildi. 80 dakikada% 5 ila% 20 ACN, 83 dakikada% 25’e, 85 dakikada% 40’a ve% 0.125’de 2 dakika boyunca% 98’e 90 dakikalık doğrusal bir gradyan 0.3 uL dakikada% 0.125 formik asit kullanıldı−1 . MS1 Spektrum orbitrap’ta elde edildi (120.000 çözünürlük, tarama aralığı 350-1.600 M/ zıplastandart AGC hedefi, otomatik maksimum enjeksiyon süresi). Şarj durumları 4-8 olan peptitler,% 21, 27 ve% 33 normalleştirilmiş çarpışma enerjisinde HCD ile parçalandı. MS2 orbitrap’ta edinildi (60.000 çözünürlük, izolasyon penceresi 1.4 M/zıpla Otomatik tarama aralığı,% 200 normalleştirilmiş AGC, 120 ms maksimum enjeksiyon süresi). Faims Pro −50, −60 ve −75 CV olarak ayarlandı.
TMT-MS verileri msconverter ile işlendi135 ve kuyruklu yıldız kullanarak arandı136 Ters diziler ve yaygın kirleticiler dahil olmak üzere insan kanonik proteomuna (uniprot Swiss-Prot 2021-11) karşı. TMEM230 varyantlarını içeren deneyler, bu tür varyantlara sahip ek bir TMEM230 dizisi de dahil olmak üzere insan kanonik proteomuna (Uniprot Swiss-Prot 2024-01) karşı arandı. Peptit kütle toleransı 50 ppm’ye ve fragman iyonu toleransı 0.02 DA’ya ayarlandı. Bu geniş kütle tolerans pencereleri, kuyruklu yıldız aramaları ve doğrusal ayrımcı analiz ile birlikte hassasiyeti en üst düzeye çıkarmak için seçilmiştir.137. TMTPRO etiketleri, lizinler ve peptid N terminal (+304.207 DA) üzerinde sabit modifikasyon, sisteinler üzerinde karboksiamidometilasyon (+57.021 DA) sabit bir modifikasyon olarak ve değişken bir modifikasyon olarak metiyonin kalıntıları üzerinde oksidasyon olarak ayarlandı. Doğrusal ayrımcı analiz yapıldı138 ve peptit spektrum eşleşmeleri (PSM’ler)% 2 FDR’ye süzüldü139. TMT-Reporter iyonları, teorik çevresinde 0.003 DA içindeki en yoğun zirveleri seçilerek ölçüldü.M/zıpla ve izotopik safsızlık için düzeltildi. Sadece tüm TMT kanallarında en az 200 toplam sinyal-gürültü oranına sahip PSM’ler ve% 50 öncü izolasyon saflığı kullanıldı140. MSSTATS kullanılarak veri özetleme, normalizasyon ve istatistikler yapıldı141–142. Peptit seviyesi normalizasyonu ve impütasyonu etkinleştirildi ve protein özetleme yöntemi, ineuronlardan endo-IP deneyleri ve diğer tüm deneyler için ‘MSSTAT’lara’ ‘logsum’ olarak ayarlandı. Önemli olarak düzenlenmiş proteinleri dikkate almak için kullanılan eşik 0.05 idiQ-i ve 1,5 kat değişiklik. PNS ve INEURONS ile endo-IP deneyleri için, durum başına üç biyolojik kopya analiz edildi (ek tablolar 4 Ve 5). Bilelerde protein IP deneyleri için grup başına dört biyolojik kopya analiz edildi (ek tablo 5), maksimum TMT kanalı sayısının sınırlaması göz önüne alındığında, iki kopya içeren bazı gruplara sahip bir veri kümesi hariç (Ek Tablo 4). Synaptik gen ontoloji zenginleştirme analizi SYNGO kullanılarak yapıldı143 (https://www.syngoportal.org/#) her deneyde tanımlanan tüm proteinleri arka plan olarak kullanarak.
DIA-MS verileri, daha önce tarif edildiği gibi DIA-NN (sürüm 1.8) kullanılarak analiz edildi.144–145. Veriler Msconvert kullanılarak MZML’ye dönüştürüldü135 Demultiplex filtresi yalnızca örtüşecek şekilde ayarlanır (10 ppm kütle hatası). Bir öncüle tam insan proteomundan (Uniprot 2022-05) spektral bir kütüphane oluşturuldu.M/zıpla350–1,050 aralığı, öncü yük 2-5 ve fragman iyonuM/zıplaAralık 145-1.450. Karbamidometilasyon, oksidasyon ve N-terminal eksizyonu modifikasyon olarak dahil edildi. Arama 10 ppm kütle doğruluğu, eşleşen koşu etkinleştirilmiş ve sağlam LC (yüksek hassasiyet) nicelleştirme stratejisi ile gerçekleştirildi. Endo-IP protokolü optimizasyon numuneleri için (Genişletilmiş Veri Şekil. 1i – l ve ek masa 1), akış aşağı analiz MS-DAP kullanılarak yapıldı146. Sadece koşul başına her üç replikatta da ölçülen peptitler (N= 3) dahil edildi. Veriler varyans stabilizasyonu normalizasyonu ve protein yöntemleri arasında mod-betons ile normalleştirildi. DEQMS algoritması, 0.01 FDR ile ayarlanmış anlamlılık eşiği kullanılarak istatistiksel analiz için seçildiP-Değer Eşiği ve Günlüğü2[fold change] 3 (Ek Tablo 1). BN birlikte fraksiyonlama deneyleri için, PCProphet ile protein kompleksi analizi yapıldı25. Üç biyolojik replikat varsayılan parametrelerle analiz edildi, sağlanan çekirdek kompleksler veritabanı olarak kullanıldı ve BN markerleri, hipotezi ortak komplekslere çökmek için kullanıldı. Daha önce tarif edildiği gibi7kompleksin her protein çiftine ko-elüsyon skorları (RF çıkış tablosundan) atandı ve akış aşağı analiz için kullanıldı. Sadece 67-kDa’da minimum pik elüsyonu ve kompleksi başına maksimum 25 proteine sahip kompleksler dikkate alınmıştır. Buna ek olarak, sadece yüksek güvene sahip aday etkileşimlerini kurtarmak için sadece iki kopyada en az 0.7 puanla etkileşimleri düşündük (Ek Tablo 2). Bu parametreler, bioplex’te bildirilen protein etkileşimlerinin optimal geri kazanımı temelinde seçildi7 (Genişletilmiş Veri Şekil. 2b, c). Corum’a dayalı seçilen protein komplekslerinin elüsyon profilleri ve Pearson korelasyon ısı haritası118 replikatlar boyunca ortalama normalize edilmiş elüsyon profili kullanılarak üretildi (aykırı değerler hariç, medyanın en farklı fraksiyonu hariç).
DSSO Çapraz Bağlantı MS verileri, Thermo Proteome Discoverer (sürüm 2.5.0.400; RRID: SCR_014477) kullanılarak XLinkx modülü ile analiz edildi.147–148. Veriler insan kanonik proteomuna (UniProt Swiss-Prot 2022-05) karşı arandı. MS2 Alım stratejisi 10 ppm öncü kütle toleransı, 20 ppm FTMS fragman kütle toleransı ve 0.6 DA ITMS fragman kütle toleransı ile seçildi. Karbamidometilasyon sabit bir modifikasyon olarak dahil edildi; Oksidasyon ve N-terminal asetilasyonu değişken modifikasyonlar olarak dahil edildi. En fazla üç tripsin yanlış öğesine izin verildi ve minimum peptit uzunluğu 5’e ayarlandı. FDR eşiği% 5’e ayarlandı ve sadece xlinkx skoru> 40 ile çapraz bağlar aşağı akış analiz için dikkate alındı (Ek Tablo 2). Çapraz bağlı tüm pozisyonların protein alanı bilgisi unIProt’tan alınmıştır (Şek. 1F) ve kopya numaraları Ref.18 (Genişletilmiş Veri Şekil. 2f). Maya iki hibrit veriler Ref.29 ve Bioplex 3.0’dan IP verileri (Ref.28; Genişletilmiş Veri Şek. 2J). BN veri kümesindeki çapraz bağlantılı protein çiftlerinin birlikte fraksiyonlanması SECAT kullanılarak değerlendirildi149. Korumdan pozitif ve negatif etkileşim ağları sağlandığı gibi kullanılmıştır. Hedef ağ, hem çapraz bağlantı hem de BN’de tanımlanan proteinler için tüm çapraz bağlanma etkileşimlerinden üretildi. Tüm hedef protein çiftleri için skorların üretilmesini sağlamak için aşağıdaki parametreler kullanıldı: tepe toplama hiçbiri, monomer eşik faktörü 1’e, minimum bolluk oranı 0’a, maksimum 48’e ve maksimum olarak ayarlandıQ-1. SECAT değeriPDeğerler, çapraz bağlantı verileri ile karşılaştırma için kullanıldı ve daha önce yukarıda tarif edildiği gibi StringDB, Corum ve Bioplex 3.0’dan bildirilen etkileşimler (Genişletilmiş Veri Şekil. 2L -n).
DHSO ve DSSO Crosslyling MS verileri Scout kullanılarak analiz edildi (sürüm 1.6.2)31. Veriler, MS’de 10 ppm hatası dahil olmak üzere varsayılan parametrelerle insan kanonik proteomuna (UniProt Swiss-Prot 2022-05) karşı arandı.1MS’de seviye ve 20 ppm hatası2seviye. Karbamidometil (kütle 57.02146) ve MMT’ler (kütle 45.987721) sırasıyla DSSO ve DHSO-çapraz bağlanmış numuneler için sabit değişiklikler olarak dahil edildi; Oksidasyon ve N-terminal asetilasyonu değişken modifikasyonlar olarak dahil edildi. En fazla üç tripsin yanlış öğütme ve iki değişken modifikasyona izin verildi ve minimum peptit uzunluğu 6’ya ayarlandı. FDR eşiği, çapraz bağlantı tiplerinin ayrılması olmadan tüm seviyelerde% 1’e ayarlandı. Aşağı analiz için ‘Kalıntı Çiftleri’ tablosu kullanıldı (Ek Tablo 2).
DMTMM Çapraz Bağlantı MS verileri PLINK2 kullanılarak analiz edildi (Sürüm 2.3.11, RRID: SCR_000084)150. Veriler, 15-PPM öncü kütle toleransı ve 20 ppm fragman kütle toleransı ile insan kanonik proteomuna (UniProt Swiss-Prot 2022-05) karşı arandı. Metilthio (C) sabit bir değişiklik olarak dahil edildi; Oksidasyon ve N-terminal asetilasyonu değişken modifikasyonlar olarak dahil edildi. Maksimum üç tripsin yanlış öğesine izin verildi ve minimum peptit uzunluğu 6’ya ayarlandı. Filtre toleransı 10 ppm olarak ayarlandı ve FDR eşiğini PSM seviyesinde% 1’e ayırdı. Filtrelenmiş çapraz bağlı bölgeler, akış aşağı analiz için kullanıldı (Ek Tablo 2). DMTMM ve DHSO çapraz bağları, her DMTMM veya DHSO çapraz bağının paylaşılan peptit sekansları nedeniyle birden fazla etkileşime uyabileceği düşünüldüğünde, DSSO çapraz bağlantıları tarafından tanımlanan tüm olası protein etkileşimlerine eşlenmiştir.
CrossLink ve BN tarafından tanımlanan tüm protein çiftlerinden bir PPI ağı oluşturuldu. Ağ başlangıçta doğal moleküler ağırlık belirteçlerinde bulunan proteinleri (BN deneylerinde referans olarak kullanılan dikenli proteinler), EEA1 (endozom afinite saflaştırması için bir sap olarak kullanılır), UBC (çoğu durumda, protein kompleksin bir üyesinden ziyade bir protein modifikasyonuna karşılık gelir) ve keratinler (yaygın olan) uzaklaştırmak için süzüldü. Ağ karakterizasyonu ve analizi IGRAPH R paketi (RRID: SCR_021238; Genişletilmiş Veri Şekil. 3A – C). Proteinler aşağıdaki ek açıklamalara göre hücre altı konuma atandı: Yukarıda açıklanan puanlama yöntemimizden endozomal proteinler (Ek Tablo 1), Golgi proteinleri (ref.140), lizozomal proteinler (Ref.151; Tablo S2 ve Tablo S12’de Ref.152), mitokondriyal proteinler (mitocarta3.0’dan (ref.153)) ve nükleer proteinler (uniprot’a dayanarak, sadece ‘çekirdek’ ve ‘kromozom’ gibi nükleer ilişkili terimlerle belirlenen proteinler). Ağ akor diyagramını (genişletilmiş veri Şekil. Şekil. 3D).
Endozomal proteinler (veya endomap.v1) etrafında merkezlenen ağ, şüpheli etkileşimlerin (yani nükleer proteinler) filtrelenmesi ve sadece endozomal proteinleri (puanlama yöntemimizle tanımlandığı gibi) ve bunların doğrudan etkileşimlerini dahil ederek üretildi. Endozomal etkileşimlerin% 8.5’ine kadar nükleer proteinler içeriyordu (Genişletilmiş Veri Şekil. 3D), sorgulanabilir olarak kabul edilebilir (bu nedenle filtrelenmiş) ve numune hazırlama ile getirilen PPI düzeyinde yanlış bağlantıyı gösterebilir. Endozomal proteinlerin ikinci dereceden etkileşimi, sadece çapraz bağlantı ile en az bir doğrudan etkileşime bağlandığında ve/veya BN tarafından iki doğrudan etkileşime bağlı olduğunda dahil edildi (Genişletilmiş Veri Şekil. 3e). Ağın çekirdek bileşeni (yani en büyük modül) Cytoscape V3.10.1 (RRID: SCR_003032) kullanılarak görüntülendi ve protein toplulukları, denetimsiz kenar-betlik analizi ile tespit edildi (Şekil. 2a). Gen ontolojisi (GO) zenginleştirme analizi, her topluluk için G: Profiler (RRID: SCR_022865) kullanılarak arka plan olarak tüm proteom (Ek Tablo 2sadece önemli GO hücresel bileşeni, GO biyolojik süreci ve en az iki protein ile Corum terimleri dahil). Komplekslere atanan proteinler arasındaki yol mesafesi analizi Corum ve Go: CC’ye dayanıyordu (sadece protein kompleksleriyle ilgili terimler; Şekil. 3b ve genişletilmiş veriler Şekil. 3f). Randomize bir kontrol olarak aynı derece dağılımı (100 permütasyon) ile grafik yeniden kablolama kullanılmıştır (Şek. 3C). Endozomal proteomun hastalık aşırı temsil analizi, skorlama yöntemimizle tanımlandığı gibi endozomal proteinler üzerinde gerçekleştirildi ve GO’da açıklandığı gibi (Go: 0005768, tarih Aralık 2024). Gen Ağı için Zenginleştirme Analizi (Disgenet)154 Doz R paketinde uygulandığı gibi gerçekleştirildi. Nörodejeneratif bozukluklar için zenginleştirme analizi, otizm spektrum bozuklukları, epilepsi ve şiddetli nörogelişimsel bozukluk ve şizofreni Ref.155ve arka plan olarak beyin eksprese edilen genlerle ClusterProfiler R paketi (RRID: SCR_016884) kullanılarak gerçekleştirildi. Nörodejeneratif bozukluklara bağlı proteinler arasındaki yol mesafesi analizi, 2.0 hastalıklarına dayanıyordu (Ref.156; 2024-02 Güncelleme; RRID: SCR_015664), Parkinson hastalığı gözden geçirilmiş genler16 ve Parkinson hastalığı genom çapında ilişki çalışmaları157–158 (Genişletilmiş Veri Şekil. 3J, K).
AF-M Colabfold V1.5.2 ile çalıştırıldı (Ref.8; RRID: SCR_025453) 40-GB A100 NVIDIA GPU’larında XL-MS ile tanımlanan tüm protein çiftleri ve endomap.v1 içindeki üç plik kombinasyonlar (toplamda en fazla 3.600 amino asit ile). AF-M sürüm 3, üç geri dönüşüm, şablon etkinleştirilmiş, bir topluluk, bırakma ve sarı gevşeme olmayan ağırlık modelleri 1, 2 ve 4 ile kullanıldı. AF-M’ye sağlanan çoklu dizi hizalamaları, MMSEQ sunucusu (RRID: SCR_022962) tarafından varsayılan ayarlarla (eşleştirilmiş + eşleştirilmemiş diziler) üretildi. SPOC ve temas siteleri daha önce tarif edildiği gibi hesaplandı30–159. Tahminlerin kalitesi, ikili tahminler için bir SPOC> 0.33 ve zamanlayıcı tahminleri için arayüz ortalama modelleri> 0.5 ile en az iki arayüz ile kabul edilebilir kabul edildi. Alphalink2 (https://github.com/lhatsk/alphalink) daha önce tarif edildiği gibi gerçekleştirildi21 intraprotein ve interplotein dsso çapraz bağlantıları kullanma. Her protein çifti için üç tahmin, farklı tohumlar kullanılarak Alphalink2 ile üretildi.
XL-MS ile tanımlanan protein çiftleri içeren tüm PDB yapıları, X-ışını ve kriyojenik elektron mikroskopi yapıları için PDB API’sının genel çözünürlükleri 70’e sahip çapraz bağlı kalıntılar dikkate alınmıştır. Çok sayıda çapraz bağlantı sunan HSP90AA1 ve HSP90AB1’i içeren çapraz bağlantılar, Alphalink2 tahminlerindeki mesafe dağılım grafiklerinden hariç tutulmuştur (Genişletilmiş Veri Şekil. 4m –) analizi tüm veri kümesinin daha temsilcisi yapmak.
MTORC1-Ragülatör kompleksinin V-ATPase ile ilişkisi, Haddock2.4 Web Sunucusu kullanılarak modellenmiştir160 (RRID: SCR_019091). ATP6V1C1-LAMPTOR2 ve ATP6V1C1-LAMPTOR4 arasında tanımlanan çapraz bağlantılar, 23 A’lik üst mesafe sınırı ve kütlesi sınırlamaları olan açık kısıtlamalar olarak kullanıldı. Tam mTORC1 -Ragülatör kompleks yapısı (PDB 7uxh)59 maksimum atom sayısındaki sınırlama nedeniyle seçilmiş V-ATPase alt birimlerine dahil edildi (PDB 6wm2 ATP6V1E1’den I ve J zincirleri, ATP6V1G1’den L ve M, ATP6V1C1’den Zincir O, ATP6V0C’den 8 ve 9 zincirleri)58. Beklenen membran topolojisi ile uyumlu en iyi skora sahip varsayımsal model seçilmiştir (Küme 5; Genişletilmiş Veri Şekil. 11b). Yapı görüntüleri Pymol 2.6.0 (RRID: SCR_000305) ile üretildi. Tüm giriş, parametre ve çıkış dosyaları Zenodo aracılığıyla şu adresten ulaşılabilir. https://doi.org/10.5281/zenodo.14679635.
Aşağıdaki yazılım, paketler ve kaynaklar ek olarak analiz ve görselleştirme için kullanılmıştır: RStudio (2023.06.0 R 4.2.1, RRID: SCR_001905 ile 421 Build 421); R paketi ggplot2 (3.5.1, rrid: SCR_014601); R paketi rcolorbrewer (1.1.3, SCR_016697); R Paketi GGREPEL (0.9.5, RRID: SCR_016223); R paketi dplyr (1.1.4); R Paket Factominer (2.11, RRID: SCR_014602); R paketi pheatmap (1.0.12, rrid: SCR_016418); R paketi factoExtra (1.0.7, RRID: SCR_016692); R Paket Proc (1.18.5); R Paketi Reshape2 (1.4.4); R Paket igraf (2.1.2); R paketi Tidyr (1.3.1, RRID: SCR_017102); R paketi LME4 (1.1.13.5, RRID: SCR_015654); R paketi GGSIGNIF (0.6.4, RRID: SCR_023047); R paketi viridis (0.6.5) (RRID: SCR_016696); Adobe Illustrator (26.5); NIAID NIH BIOART SAYFASI.
Örnek boyutu, kopya sayısı ve istatistiksel testler, şekil efsanelerinde ve karşılık gelen bölümlerinde belirtilmiştir. Yöntem. Şekil 2’de validasyon ve temsili deneyler 3d, H ve genişletilmiş veri incir. 5c Ve 7g bir kez gerçekleştirildi, genişletilmiş veriler incir. 5e, h, k, m Ve 8c iki kez gerçekleştirildi ve Şekil. 4G ve genişletilmiş veriler Şekil. 7f bağımsız deneylerde benzer sonuçlarla üç kez gerçekleştirildi.
Araştırma tasarımı hakkında daha fazla bilgi,Doğa portföyü raporlama özeti Bu makaleye bağlı.
 1
Küresel ısınma sayesinde toz fırtınaları için mükemmel fırtına
38 kez okundu
1
Küresel ısınma sayesinde toz fırtınaları için mükemmel fırtına
38 kez okundu
 2
Yok olan bir savanın ortasında, yeni koridor vahşi yaşam için ‘büyük bir kazanç’
14 kez okundu
2
Yok olan bir savanın ortasında, yeni koridor vahşi yaşam için ‘büyük bir kazanç’
14 kez okundu
 3
Küçük bir ada sürdürülebilirlikte büyük dersler sunar
14 kez okundu
3
Küçük bir ada sürdürülebilirlikte büyük dersler sunar
14 kez okundu
 4
Gen Z, ağaçlıklarla koruma çağrısına liderlik ediyor
13 kez okundu
4
Gen Z, ağaçlıklarla koruma çağrısına liderlik ediyor
13 kez okundu
 5
Hedefsiz varyasyonlar: Mayıs 2025
12 kez okundu
5
Hedefsiz varyasyonlar: Mayıs 2025
12 kez okundu