
C. sativa Genetik çeşitliliği, tarihi ve agronomik değeri en üst düzeye çıkarmak için pangenom örnekleri birden fazla kaynaktan seçildi. Pangenomun büyük bir kısmı, elit çeşitleri içeren Oregon CBD (OCBD) üreme programından gelir; 1970’lerden günümüze gelen temel esrar çizgileri; ve üreme programının farklı yönleri için kullanılan elit üçlüler (Genişletilmiş Veri Şek. 1 Ve 2Ek masa 1 ve Ek Şek. 1). Kalan çeşitler ABD Tarım Bakanlığı (USDA) Germplazm Kaynak Bilgi Ağı (GRIN) ve Alman Federal Genebank (IPK Gatersleben) depolarından ve çeşitli yetiştiricilerden Salk Enstitüsü tarafından yapılan koleksiyonlardan geliyor. Pangenom, Avrupa ve Asya lifi ve tohum kenevir, vahşi popülasyonlar, Kuzey Amerika esrar (tip I) ve Kuzey Amerika yüksek kannabinoid verimi (CBD veya CBG) kenevir (tip III ve IV) içerir. Ek kannabinoid çeşitliliği, CBD veya THC’nin yüksek pentil veya propil (varin) homologları ve kannabinoid içermeyen (tip V) bitkilerin ekspresyonu gösteren kemotiplerle temsil edilir. Çiçeklenme süresi varyasyonu, hem normal kısa gün hem de gün bağımsız (otomatik işgücü) fenotiplerin dahil edilmesiyle yakalanır (Ek Tablo 1).
EH23A (HO40) ve EH23B (ERB), bir F, F, bir F, bir1 Ebeveynler, ERB ve HO40 arasındaki bir haçtan kaynaklanan her ikisi de OCBD’den tescilli kadın inbred çizgileri. ERB, Avrupa HC Kenevir ile daha yakından ilişkili ilaç tipi grubun bir parçası olan bir DN (otomatik işleyici), tip III (CBDA-dominant) bitkisidir. HO40, tip I propil kanabinoid (THCVA ve THCA) üreten, kısa günlük çiçeklenme duyarlıdır ve Asya kenevirine daha yakın bir afiniteli ilaç tipi esrar grubunun (MJ) bir parçasıdır. Genetik olarak dişi (XX) ERB bitkisi, gümüş tiyosülfat ile tedavi ile erkek çiçekler üretmeye teşvik edildi ve HO40’ı tozlaştırmak için kullanıldı. F’den bir kişi1 Genom sekanslaması için popülasyonlar (erbxHO40_23) seçildi. Akış sitometrisi kullanılarak ERB × HO40_23’ün başlangıç genom boyutu tahminleri 1445.6 mb’lik bir diploid genom boyutunu tahmin ediyor (722.8 mb haploid genom boyutu). Yaprak dokusundan yüksek moleküler ağırlık (HMW) DNA ekstrakte edildi. DNA ekstraksiyonu ve kütüphane hazırlığını takiben (bkz. ‘HMW DNA izolasyonu ve genom sekanslama’) HIFI okumaları Pasifik Bioscience (Pacbio) devamı II’de üretildi. Hifiasm v0.16.159 daha sonra başlangıç montajlarını üretmek için Hi-C okumaları ile birlikte kullanıldı. Montajdan sonra, Hi-C okumaları, meyve sıkacağı v1.6.2 boru hattı kullanılarak HiFiasm_hic contig’lerine hizalandı60 3D-DNA boru hattının 180922 sürümünü kullanan sipariş ve oryantasyon izleme61. İskele montajları daha sonra Juicebox v1.11.08 kullanılarak manuel olarak düzeltildi62.
Yukarıda açıklanan tüm genom dizileme verilerine ek olarak, erbxho40_23, bir F oluşturmak için seçkin çiçeklerin maskülizasyonu kullanılarak kendi kendine tozlaştı.2 Mapping popülasyonu. Bu f’den2 Nüfus, bireyler otoflower ve varin içeriği için puanlandı ve Nrgene (Nrgene Technologies) tarafından Illumina 100 bp okumaları kullanılarak dizildi. Illumina WGS genotipleme çalışmaları, bu popülasyondan 288 bitki ve ayrıca ErbxHO40_23 ebeveyni üzerinde gerçekleştirildi. Trim_galore, dizileri aşağıdakiler kullanarak düzeltmek için kullanıldı: –2 renk 20, analiz için 271 kişiye neden oldu63. Ortalama numunelerde 8.5 × kapsama alanı vardı. Her bir örneği eh23b.softmasked.fasta’ya hizalamak için minimap kullanıldı. Freebayes varyantları çağırmak için kullanıldı: -g 4500 -0 -n 4 –trim-complex-tail–din-alternate-count 364. BCFTOOLS, Qual> 20 skorunu filtrelemek için kullanıldı (% 99 şans varyantı var)65. Son olarak, Vcftools66 Araçlar daha sonra SNP’leri daha fazla filtrelemek için kullanıldı: —semove-indels —mingq 20 —maf 0.25-Maxming 1–min-alleles 2 —max-alleles 2 —stdout –recode66; ErbxHO40_23 örneğinde sadece heterozigot (0/1) olarak puanlanan alanlar tutuldu, bu da 93.251 SNP ile sonuçlandı.
Yüksek performanslı sıvı kromatografisi (HPLC), daha önce iyice tarif edilen protokole göre gerçekleştirildi67 F dahil bu çalışmada kullanılan tüm bitkilerde göreceli propil ve pentil kanabinoid içeriğini belirlemek için2 döl. Kısacası, her bir bireyden olgun çiçek dokusu toplandı, −80 ° C’de donduruldu ve kanabinoidler metanol içinde ekstrakte edilmeden önce homojenleştirildi.
ERBXH040-21 fideleri kontrollü çevresel koşullar altında yetiştirildi. Erken ve geç çiçekler, yeşillik, 12 saatlik endüktif ışık rejimi, kökler ve sürgün ipuçları altında yapraklar dahil olmak üzere bitkilerin gelişimi sırasında çeşitli dokular toplandı. Toplam RNA ekstraksiyonu, üretici protokollerini takiben Qiagen RNeasy Plus kiti kullanılarak yapıldı. Toplam RNA, quit RNA analizi ve Tobestasyon 4200 kullanılarak ölçüldü. Kütüphane hazırlığından önce DNaz tedavisi, ardından ampure boncuk temizleme ve HMR rRNA tükenmesi yaptık. Kütüphane hazırlığı, üretici protokollerini takiben NebNext Ultra II RNA Kütüphane Hazırlık Kiti ile yapıldı. Daha sonra bu kütüphaneler 2 × 150-bp konfigürasyonunda NovaseQ6000 platformunda çalıştırıldı.
Somon v1.6.0 kullanarak gen ekspresyon seviyelerini ölçtük68. Kısacası, sekanslamadan elde edilen ham eşleştirilmiş son okumalar, her iki haplotipten (EH23A ve EH23B) CDS’lere eşlenmiştir ve bolluk, aşağı akış analizleri için milyon başına transkriptlerde (TPM) tahmin edilmiştir. Haritalama oranları Samtools Flagstat ile hesaplandı65. Belirli bir gen için minimum TPM eşiği ≥0.1 idi. Haplotip gen çiftleri, Blastp ve McScanx kullanılarak karşılıklı en iyi hit ve synteny ile tanımlandı69ve sadece her iki haplotip arasında paylaşılan genler dahil edildi. Minimum ≥% 95 sekans benzerliği ve haplotipler arasında 5 tpm fark eşiği uygulandı. Görselleştirme, matplotlibin bir kombinasyonu kullanılarak yapıldı70Scipy71 ve numpy72ve ifade değerleri ısı haritalarında günlük olarak gösterilir2TPM log kat değişimini temsil etmek için. Biyolojik Süreçlerin Zenginleştirilmesi GO terimleri Topgo ile gerçekleştirildi73 Aşağıdaki parametrelerle: sonuç ağırlığı <- runtest (topgodata, algoritma = “ağırlık01”, istatistik = “fisher”). Aşağıdaki komutla çoklu test düzeltmesi gerçekleştirildi: FullResults $ p.adj <- p.Adjust (AS.NUMERIC (FullResults $ Weightfisher), Metod = “FDR”). Arka plan gen evreni, EH23A veya EH23B'den bir GO terimi olan tüm genleri içeriyordu.
Aynı gelişim zaman noktasında, 08:00 ve 20: 00’de toplam 16 örnek için dört as yüksek bitkisinden çiçek ve yaprak dokusu topladık. Ace yüksek erkekler, normal dış mekan koşulları altında dişi bitkilerden birkaç hafta önce çiçek açtığından, bitkiler uzun günlerde çimlenmiştir ve büyütülmüş ve çiçeklenme için endüktif kısa gün koşullarına aktarılmıştır, bu da hem erkek hem de dişi bitkilerin aynı anda çiçek geliştirmesine neden olmuştur. Sirkadiyen veya günlük ifadelerinden bağımsız olarak tüm transkriptleri yakalamak için örnekler günün iki kez toplandı.74. RNA, Qiagen Bitki RNA kiti ile ekstrakte edildi. Kütüphane hazırlığı Oxford Nanopore Technologies (ONT) tam uzunlukta cDNA kiti ile gerçekleştirildi. Tam uzunlukta cDNA’yı haplotip ile çözülmüş Ace High (AH3A/B) genomlarına minimap2 ile hizaladık (v2.24)75 ve gen ekspresyonu somon v1.6.0 kullanılarak ölçüldü68. Tüm dokuya özgü erkek ve dişi örnekler (iki erkek bitkiden (A ve B bitkileri, 08:00 ve 20:00) (08:00 ve 20:00) (C ve D bitkileri 08: 00 ve 20:00’de toplanan)) cinsiyete dayalı ekspresyon atandı. Her cinsiyete özgü dokuda dört kopyası vardı (örneğin, iki farklı zaman noktasında iki erkek bitkiden örneklenen erkek çiçeklerden gen ekspresyon ölçümleri ortalaması alınmıştır). İki önyargılı ekspresyon kategorisi tanımlanmıştır: birincisi, erkek veya dişi örneklerde, diğer cinsiyete göre daha yüksek (en az 5.0 tpm daha büyük) ortalama ifade (en az 5.0 tpm daha büyük); ve ikincisi, genlerin bir cinsiyette eksprese edilmediği (tüm kopyalar için 0.0 tpm), ancak diğer cinsiyette ortalama en az 1.0 tpm ekspresyonu vardı. Topgo ile GO Dönem Analizi için73her iki önyargılı gen ekspresyonu kategorisi birleştirildi. Tamamen sintenik genler, Genespace kullanılarak X ve Y kromozomları (AH3MA/B, BCMA/B, GRMA/B ve Kompa/B) ile dört genom setinde tanımlandı ve PAR, SDR veya X-spesifik bölgedeki yere göre gruplandırıldı.
Dovetail Omni-C kütüphanesi için, kromatin çekirdekte formaldehit ile yerinde sabitlendi ve daha sonra çıkarıldı. Sabit kromatin DNase I ile sindirildi, kromatin uçları onarıldı ve biyotinile edilmiş bir köprü adaptörüne bağlandı ve ardından adaptör içeren uçların yakınlığı. Yakınlık ligasyonundan sonra çapraz bağlar tersine çevrildi ve DNA saflaştırıldı. Saflaştırılmış DNA, ligated fragmanların içi olmayan biyotini uzaklaştırmak için tedavi edildi. Sekans kütüphaneleri, NebNext ultra enzimler ve Illumina uyumlu adaptörler kullanılarak üretildi. Biyotin içeren fragmanlar, her kütüphanenin PCR zenginleştirilmesinden önce streptavidin boncukları kullanılarak izole edildi. Kütüphane, ~ 30 × dizi kapsamı üretmek için bir Illumina HiSeqx platformunda sıralandı. Sonra kullanılır (yukarıdaki okuma çifti bkz.) MQ> 50 iskele için okur. Ek Hi-C kütüphaneleri Faz Genomics Proximo Hi-C kiti (Plant) sürüm 4 kullanılarak üretildi.
Tüm numuneler bir Pacbio devam filmi II’de dizildi. ‘Michael’dan kaynaklanan örnekler için (Ek Tablo 1), HMW DNA, ‘Bitki Yaprağı GDNA’ ONT protokolünde tarif edildiği gibi Carlson lizis tamponu ve Qiagen genomik ipuçları kullanılarak izole edildi. Arabidopsis yöntem. DNA, ONT kısa fragman eliminasyon kiti (EXP-SFE001) kullanılarak 10-25 kb’den daha uzun fragmanlar için daha fazla seçildi. HMW DNA daha sonra gobestasyon genomik DNA ekran (Agilent 5067-5365) veya FEMTO puls genomik DNA 165 kb kiti (Agilent FP-1002-0275) ile doğrulandı. ‘OCBD’den kaynaklanan numuneler için (Ek Tablo 1), HMW DNA modifiye edilmiş bir protokol kullanılarak izole edildi76. Kısacası, numuneler bir harçta öğütülmüş ve sıvı azotlu havaneli, iki kloroform: izoamil yıkama döngüsü gerçekleştirildi ve orijinal protokolün yerine toplam saf NGS boncukları (Omega Biotek) kullanıldı. Genomik DNA (GDNA) kalitesi ve saflığı daha sonra kütüphane hazırlığına başlamadan önce bir nanodrop olan (Thermofisher) kullanılarak değerlendirildi. Sürekli uzun okuma (CLR) kütüphaneleri PACBIO PN 101-693-800 V1 protokolü kullanılarak yapıldı. GDNA üzerindeki boyut seçimleri, 60-90 kb’lik fragman dağılımları üretmek için 30-40 kb taban çifti ile 30-40 kb baz çifti ile mavi Pippin U1 Yüksek Geçiş 30-40 kb kaset kullanılarak yapıldı. HIFI dairesel konsensüs sekanslama (CCS) kütüphaneleri PacBio protokolüne (PN 101-853-100 V5) göre hazırlandı. Modal pik ~ 18 kb ile kesilmiş gDNA fragman dağılımları, 10 kb’nin altındaki her şeyi çıkarmak için Covaris ve Blue Pippin S1 High Pass 6-10 kb kaset kullanılarak üretildi.
Hifiasm_hic, HiFiasm_trio_ragtag, HiFiasm_ragtag ve HiFiasm etiketli tüm genomlar (Ek Tablo 1) hifiasm v0.16.1 kullanılarak monte edildi59. Mevcut olduğunda, Hi-C veri ve HIFI ebeveyn üçlü verileri de sırasıyla HiFiasm_hic ve HiFiasm_trio_ragtag tiplerini tanımlayan montaj işlemine dahil edildi. CLR montajları Pacbio Smrt Tools 9.0 Suite’ten Falcon Unzip kullanılarak üretildi77 ve CCS etiketli genomlar hicanu v2.2 ile birleştirildi78. Montajdan sonra, Hi-C okumaları, meyve sıkacağı v1.6.2 boru hattı kullanılarak HiFiasm_hic contig’lerine hizalandı60 3D-DNA boru hattının 180922 sürümünü kullanan sipariş ve oryantasyon izleme61. İskele montajları daha sonra Juicebox v1.11.08 kullanılarak manuel olarak düzeltildi62. Hifiasm_ragtag ve hiFiasm_trio_ragtag düzenekleri, 24 Hi-C iskele genomlarının bölünmüş kromozomları kullanılarak iskele edildi ve Yak-0.1 (github.com/lh3/yak) ile kontrol edildi. Sourmash v4.6.179 kromozomlar ve her skafçoz eklemesi arasında bir Jaccard benzerlik matrisi oluşturmak için kullanıldı ve kromozom 1 ila x’in en benzer versiyonu, ragtag v2.1.0 ile iskele için bir referans oluşturmak üzere birleştirildi.80. Benzerlik matrisi Y kromozomunu en iyi eşleşme olarak tanımlarsa, montaj yerleşimsiz kaldı. Busco v5.4.379 Eudicots_odb10 veri kümesi ve montaj-stats v1.0.1 (https://github.com/sanger-pathogens/assembly-stats) tüm montajlarda bütünlüğü ve bitişikliği ölçmek için kullanılmıştır.
78 iskele ve yumuşak maskeli montajların tümü grafik pangenomu, minigraph-caktus ile üretildi20. Bir AppTainer (v1.1.8) görüntü içinde kaktüs-pangenom komutunu kullandık81 (https://quay.io/comparative-genomics-toolkit/Cactus:v2.6.7-gpu) ve aşağıdaki parametre bayrakları:-Referans EH23A EH23B –VCF –vcfreference EH23A EH23B —giraffe –chrom-og –chrom-vg –viz –gfa-gbz. Seqfile girişinin yanı sıra çeşitli formatlardaki çıkış grafiği (VG, PAF, HAL vb.) https://resources.michael.salk.edu. Ayrıca, minigraph-cactus GFA çıkışından VCF dosyalarını türetmek için VG Deconstruct -a -C (VG Tools v1.61.0 “PLODIO”) kullanarak ve daha sonra VCFBUB-Max-Ref-Longth 100000-Max-Longs to Long to KB’yi kullanarak her montajın koordinatları açısından Pangenom boyunca varyantları derledik (bkz. 78CSathaps_MinigraphCactus_.vcf.gz)20–82–83.
Her bir PGGB grafiğinin iki sürümünü oluşturduk, biri ‘montaj dosyaları’ tablosunda ve jbrowse örneğinde sağlanan fasta dosyaları ile https://resources.michael.salk.edu (karışık yönlendirme) ve dizilerin sürekli olarak EH23A’da (tutarlı yönlendirme) karşılık gelen homolog kromozomun artı ipliğine uyacak şekilde yönlendirildiği fasta dosyaları ile.
PGGB grafiği 16CSatasms için, aşağıdaki 16 iskele ve yumuşak maskelenmiş düzeneklerden otozomal kromozom başına bir grafik ürettik: AH3MA, AH3MB, BCMA, BCMB, EH23A, EH23B, GRMA, GRMB, KCDV1A, KCDV1B, KCDV1A, K2A, SAN2A, SAN2, YMV2A. PGGB için giriş olarak kromozom başına bir kombine fasta dosyası oluşturduk (bkz. 16csatasms_chr[1-9]_combined.fa.gz ve 16csatasms_chr[1-9]-oorient_combined.fa.gz Sırasıyla Tutarlı ve Karışık Oryantasyon fasta girişleri için, Resources.Michael.Salk.edu). Bu boyutta ve tekrarlayan içerikteki genomları analiz etmek için hesaplama gereksinimleri nedeniyle birleştirilen tüm düzeneklerin tamamı için tek bir grafik yerine kromozom grafikleri oluşturduk (Genişletilmiş Veri Şekil. 6).
PGGB grafiği 13csatsexchroms, 13 iskele ve yumuşak maskelenmiş seks kromozom sekansları Ah3ma.chrx, ah3mb.chry, bcma.chrx, bcmb.chry, eh23a.chrx, grma.chry, grmb.chrx, kcdv Kompa.chrx, Kompb.chry, San2a.chrx ve San2b.chrx bir fasta dosyasında birleştirildi (bkz. https://resources.michael.salk.edu).
NextFlow v24.04.3.591684 NF -Core/Pangenome v1.1.2 – CanGuro Dağıtımını çalıştırmak için kullanıldı85–86 Pggb22 NextFlow Tekillik Profili içinde. Grafik oluşturma için tüm varsayılan PGGB ayarları kullanıldı. PGGB grafiği 13CSatsexchroms için, her bir montajın koordinatlarına göre panenom boyunca sekans varyasyonunu derlemek için-flag –vcf_spec, vcfbub —max-ref-uzunluk 100000-Max-Level 0 ile daha fazla işlendi ve bu> 100 kb ile düzlemli ibre varyantları çıkardı ve çıkardı.20 (bkz..vcfbub.vcf.gz ve 13csatsexchroms_pggb-oorient_VCF’ler için .vcfbub.vcf.gz Dosyalar, sırasıyla tutarlı ve karışık yönelimli giriş fastas ile oluşturulan grafiklerden https://resources.michael.salk.edu). PGGB grafiği 16CSatasms için PGGB, –VCF_SPEC bayrağı olmadan çalıştırıldı ve bunun yerine VG dekonstruct -a -a, her bir otozomal kromozom için son GFA dosyasından panjenom boyunca dizi varyasyonunu derlemek için kullanıldı (VG Tools v1.61.0 “plodio”)82–83. Autozom başına VCF dosyaları, BCFTOOLS kullanılarak her montaj için tek bir dosyada birleştirildi.65 ve daha sonra iç içe varyantları düzleştirmek ve 100 kb uzunluğunda olanı kaldırmak için VCFBUB-MAX-REF-RENLART 100000-MAX-LEVEL 0 ile işlenmiştir.20 (Bkz. 16CSatasms_PggbbyChrom_.vcf.gz ve 16csatasms_pggbbyoriginalchrom_.VCF.GZ, Tutarlı ve Karışık Oryantasyon Giriş Fastas ile oluşturulan grafiklerden VCF’ler için Sırasıyla Resources.Michael.Salk.edu). Tutarlı ve karışık yönelimli girişlerle üretilen her grafik çifti için aynı parametreler kullanıldı.
Grafik pangenomlarının görselleştirmeleri, tutarlı yönlendirme girişi fastas üzerinde çalıştırılan PGGB boru hattının final_gfa dosyalarından oluşturuldu. VG dosyaları VG Convert kullanılarak GFA dosyalarından türetildi82–83. Daha sonra prepare_vg.sh ve prepare_chunks.sh, sekans tüpü harita sunucusunun yerel bir örneğinde ilgilenilen bölgelerdeki pangenom varyasyonunu görselleştirmek için kullanıldı (https://github.com/vgteam/queencetubemap.git4 Eylül 2024’te klonlanmıştır).
EH23 F’den kısa okuma dizileri2 Nüfus ve Ren ve ark.2 VG Graffe ile Pangenom Grafiğine hizalandı (Örnek Komut: VG Giraffe -z {Input.Inputgbz} -d {Input.Inputdist} -m {Input.inputmin} -f} -tpputr2} -t {input.input.inputr2} -t {input.utput.utpput.input.inputr287. Özet istatistikler VG istatistikleri ile toplandı82 (Örnek komut: vg istatistikleri -a {input.inputgam} {input.inputgbz}> {output.utputFile}). VG Pack ile GAM dosyasından okuma desteğini hesaplayın82 (Örnek komut: vg paketi -x {input.inputgbz} -g {input.inputgam} -q 5 -t {Threads} -o {output.utputFile}). F için varyantlar2 VG çağrısı ile haritalama popülasyonu çağrıldı88 (Örnek komut: vg call -gbz {input.inputgbz} -k {input.inputpack} -s eh23b -t {Threads}> {output.utputFile}). VCF dosyalarının aşağı akış işlemesi bcftools ile gerçekleştirildi65 (Örnek Komutlar: (1) Bcftools Görünümü -a -f Geçiş Birleştirilmiş.sorted.vcf.gz> birleştirilmiş.sorted.a.pass.vcf.gz; (2) Bcftools normu -fasta-ref eh23b.softMasked.fasta -m -Ayged.Sorted.Vass.vcf.gz> Birleştirilmiş.sort.a.pass.normed.vcf.gz (3) Bcftools normu. Doğrusal referans tabanlı VCF dosyasıyla karşılaştırmak için pangenom grafik tabanlı VCF dosyasının filtrelenmesi VCftools ile gerçekleştirildi66 (Örnek Komut: Vcftools-Remove-indels —mingq 20 —maf 0.25-Maxming 0.3–Min-Alleles 2 —max-Alleles 2-Stdout —recode —gzvcf birleştirilmiş.sorted.a.pass.normed_no_dups.vcf.gz.gz> birleştirilmiş.sorted.a.pass.normed_no_dups.more_filter_missing0.3.vcf.gz).
Yukarıda açıklanan grafik pangenomları için giriş ve çıkış dosyaları (minigraph-cactus tarafından üretilen 78CSatap ve PGGB tarafından üretilen 16csatasm ve 13csatSexchrom’lar) https://resources.michael.salk.edu. VCF dosyaları, Esrar Genomlar Jbrowse örneği https://resources.michael.salk.edu.
Genomic, oluşturulan RAW ONT FAST5 dosyalarından okurlar Esrar Sekans örnekleri metilasyon çağrısı için kullanıldı. Aynı bireyler için üretilen genom montajları hizalama referansları olarak kullanılmıştır. FAST5 verileri, POD5 yazılım paketi kullanılarak POD5 formatına dönüştürüldü (https://github.com/nanoporetech/pod5-file-format). Metilasyon çağrısı ONT taban çağrısı yazılımı Dorado sürüm 0.3.4 (https://github.com/nanoporetech/dorado/). Dorado, Metillenmiş sitozinleri tanımlamak için ham POD5 verilerini ve referansını kullanır. Bu, her bir çizgi için sekanslama koşullarına göre, R9.4.1 veya R10.4.1 gözenek tipi ve 400 bps translokasyon hızı için eğitilmiş süper yüksek doğruluk (SUP) taban çağırma modeli ile gerçekleştirildi. Her bir numuneden üretilen monte edilmiş genomlar, 5MC ve 5HMC metilasyon çağrıları içeren mm/ml etiketlerle hizalanmış bir BAM dosyası oluşturmak için referans olarak kullanıldı. Bunlar daha sonra modkit ile yığıldı (https://github.com/nanoporetech/modkitve tüm CG bölgelerindeki genom çapında metilasyon frekanslarını hesaplamak için kazınmış çağrılar (5HMC ile 5MC ile toplama) kullanıldı.
Gen modeli tahmini çok aşamalı bir boru hattı içeriyordu ve tüm montajlara uygulandı.
İlk olarak tekrarlama kütüphanesini tekrarladık89 Az sayıda yüksek kalitede Esrar montajlar ve önceden var olan tekrar kütüphaneleri. Orthofinder kullandık (v2.5.4)90 tekilleştirme için tekrarları gruplamak. Son tekrar kütüphanesi, 5.793 gruptan toplam 6.262 sekans için her bir tekrar ortogruptan (minimum 1 sekans) dizilerin% 10’unu içeriyordu.
Finola (GCA_003417725.2)
CBDRX (GCF_900626175.2)
Purple_kush (GCA_000230575.5)
Erbxho40_23
Erbxho40_23
İ3
JL (GCA_013030365.1)
ERB_F3
Cannbio-2 (GCA_016165845.1)
W103
Jl_mother (GCA_012923435.1)
FB30
TS1_3_V1
HO40
Tüm 193 genom için tekrarlar tekrarlamacı ile maskelendi (v4.1.2)91 tekrar kütüphanesini kullanarak (yukarıda).
Tsebra boru hattı ile gen modellerini tahmin ettik (fren v2.1.6 kullanarak)92. Tsebra’yı çalıştırmak için bir Snakemake iş akışı geliştirdik, burada bulunabilir: https://gitlab.com/salk-tm/snake_tsebra. Esrar ve diğer organizmalardan önceden var olan çeşitli protein kütüphanelerini kanıt olarak dahil ettik: (a) Arabidopsis thaliana; (B) Thebroma kakao; (C) G. Max; (D) Rhamnella Headworks; (e) Ziziphus jujuba; (F) Doğu; (G) Vine Vinifera; (H) Prunus persica; (Ben) Daha dikkat çekici; (J) C. sativa; (k) H. Lupulus.
RNA-seq kütüphaneleri (Ek Tablo 2) Hisat2 ile hizalandı (v2.2.1)93 Kısa okunan eşleme veya minimap2 için (v2.24)75 tam uzunlukta cDNA için. Kısa okunan Illumina verileri fastp ile kesildi94. İfade verileri, gen modeli kanıtı olarak Tsebra boru hattına dahil edildi.
Gen modellerinin varsayılan fonksiyonel ek açıklamaları, Eggnog-Mapper (v2.0.1) kullanılarak atandı.95.
Genel gen modeli kalitesi ve eksiksizliği, genom busco (v5.4.3) karşılaştırılarak değerlendirildi.96 EUDICOTS_OCDB10 veri kümesindeki proteom Busco skorlarına skorlar (Ek Tablo 1: https://doi.org/10.6084/m9.figshare.25869319.v2).
EDTA v1.9.697 ayrıca esrar pangenomundaki TE’leri aşağıdaki komutla tanımlamak için kullanılmıştır: edta.pl – -genome {inputfastafile} –anno 1 -threads 32.
78 kromozom düzeyinde, haplotip aşamalı genomlar için her kromozom çifti için ideogramlar, GGPLOT2 kullanılarak oluşturuldu. [https://ggplot2.tidyverse.org] R’de (www.r-project.org) (İncir. 1 ve genişletilmiş veriler Şekil. 5). Her kromozomun uzunluğu ‘nuccomp.py’ kullanılarak belirlendi (https://github.com/knausb/nuccomp) ve grafiği başlatmak için ggplot :: geom_rect () ile kullanılır. Her kromozom için bir milyon baz çifti penceresi oluşturuldu, burada CPG motiflerinin sayısı, her pencere için motif_counter.py programı ile sayıldı (https://github.com/knausb/nuccomp). CPG sayısı, pencere boyutuna bölünerek bir hıza dönüştürüldü; Bu aynı zamanda bir milyondan daha az boyutta olan her kromozomun son penceresini de barındırdı. Bu oranlar, minimum oranın çıkarılması ve daha sonra kromozom bazında maksimum oran (minimum oranın çıkarılmasından sonra maksimum oran) ile bölünmesiyle ölçeklendirildi. CPG motifinin sentromerik bölgedeki zenginleştirilmesini görsel olarak vurgulamak için, bir tane alınarak ve her pencere için CPG oranının çıkarılmasıyla CPG oranının bir tersi alınmıştır. Bu ölçekli, ters CPG hızı, her bir MBP penceresinin genişliği için kullanıldı ve viridis magma paleti kullanılarak gen yoğunluğuna göre renklendirildi (https://doi.org/10.5281/zenodo.4679424).
Her kromozom çifti arasındaki yapısal varyasyon, minimap2 kullanılarak belirlendi75 hizalamalar. Minimap2 karşılaştırmaları Syri kullanılarak açıklandı98. Syntenous ve ters bölgeler, GGPLOT2 :: geom_polygon () kullanılarak PLOTSR’den esinlenen bir şekilde çizildi99 ancak R’de uygulanmıştır (github.com/vininglab/cannabishangenome).
EH23 Haplotipleri A ve B içindeki aday lokuslarının yeri Blastn kullanılarak belirlendi100. Sorgu dizileri aşağıdaki gibidir: CBCA sentaz (Ly658671.11), CBDA sentaz (AB292682– AB292683– AB292684), Thca sözde (AB212829– AB212830) ve Oliveololic Asit Siklaz (NC_044376.1C4279947-4279296, NC_044376.1: C4272107-4271242). Bu diziler, blastn_queries_rrna_cann.fasta dosyasındaki sentromerik, telomerik ve rRNA dizileri ile birleştirildi (https://github.com/vininglab/cannabishangenome). Blastn aşağıdaki seçeneklerle çağrıldı: -task megablast -yvalue 0.001 -PERC_Idendent 90 -QCOV_HSP_PERC 90. Tablo sonuçları (konu kromozomu, hizalama, hizalamanın konusu sonu) R’ye okundu ve ggplot2 ile ggplot2 ile çizildi::https://ggplot2.tidyverse.org).
ONT ve PACBIO tabanlı uzun okuma bazlı genom düzenekleri101. Centromerler, değiştirilmiş ayarlar (1 1 2 80 5 2000 -D -H) kullanılarak tandem tekrar bulucu (TRF; V4.09) kullanılarak genomların arama yapılarak tanımlandı.102. Tandem tekrarları, sentromerleri tanımlamak için önceki yöntemlerimize göre en yüksek kopya sayısı tandem tekrarını bulmak için yeniden biçimlendirildi, toplandı ve çizildi.101 (Genişletilmiş Veri Şekil. 5c).
Telomerler iki farklı yöntem kullanılarak tahmin edildi. Birincisi, TRF çıkışı, kanonik telomer tabanı tekrarının 14 farklı versiyonu için 7 dönem ile tekrarlar için sorgulandı: AAACCCT, AACCCTA, ACCCTAA, CCTAAC, TAAACCC, TTTAGGG, TTAGGTTA, GTGTTAG (GTGTTAG ( -a ‘periyodu = 7’ *.Genome.fasta.1.2.80.5.200.2000.dat.gff | ‘Konsensüs = aaaccct | konsensüs = aacccta | konsensüs = Accctaa | konsensüs = ccctaaa | konsensüs = CCTAAAC | konsensüs = ctaaacc | konsensüs = taaaccc Consensus = tttaggg | konsensüs = ttagggt | konsensüs = tagggtt | konsensüs = agggttt | konsensüs = gggttta | konsensüs = ggtttag | konsensus = gttTagg ‘ -). İkincisi, telonum algoritmamızı kullanarak RAW ONT ve Pacbio’nun telomer dizilerini okuduklarını araştırdık.103. Sonuçlar pangenom düzeneklerinde değişken olmasına rağmen, genel olarak, kromozomun sonunda Pacbio düzenekleri için ortalama 16 kb ve ONT düzenekleri için 60 kb olan telomer sekansı bulundu. ONT ve PACBIO telomer uzunluğu arasındaki farklar büyük olasılıkla giriş okuma uzunluğunu sırasıyla> 100 kb ve 15-20 kb yansıtmıştır. Ham okumalarının telonum analizi, gerçek boyuttan daha kısa iken, telomer dizisine sahip çoğu kromozomla tutarlı montajlardan dağılımları destekledi. Esrar Telomerler bir eudicot için daha uzun taraftadır ve tıbbi kullanımlar için ağırlıklı olarak klonal yayılımı ile açıklanabilir.104.
Centromer dizisi, genomlarda daha yüksek dereceli tekrar (HOR) yapısına sahip en bol tekrar olacağı hipotezine göre tanımlanmıştır.101–105. Pacbio hifiasm düzenlemelerinde HOR ile iki farklı tekrar tanımlandı, oysa ONT düzenlemelerinde ve ONT dizisine dayanan önceki CBDRX düzeneğinde sadece bir tane bulundu.11. En yüksek kopya sayısı tekrar, 20-30 MB (toplam genomun% 2-4’ü) arasında değişen 370 bp idi, 740 ve 1.110 bp’de HOR (Genişletilmiş Veri Şekil. 5). İkinci en yüksek ve ONT düzeneklerinde bulunan tek kişi, 3-5 MB (toplam genomun% 0.4-1.0’ı) arasında değişen ve 474 ve 711 bp’de HOR (Genişletilmiş Veri Şekil. 5). 370-bp tekrarının kromozomla çözülmüş genomlara haritalanması, bu tekrarın öncelikle telomer dizisinin yanındaki kromozomların sonunda bulunduğunu ortaya koydu, bu da CS-1 alt-telomerik tekrarıyla ilişkili olabileceğini öne sürdü106. Varsayılan 370-bp sentromerik tekrarının ve CS-1 alt telomerik tekrarının karşılaştırılması aynı tekrar öğesi olduğunu gösterdi. Aksine, varsayılan 237-bp sentromerik tekrar ağırlıklı olarak Chr. 6 ve Chr. 8 Tahmin edilen sentromer bölgesinde (Şek. 1A ve genişletilmiş veriler Şekil. 5). Bununla birlikte, öngörülen sentromer bölgesindeki (CPG, metilasyon, gen içeriği ve TE’lere dayalı) montajlar boyunca tüm kromozomlarda daha küçük 237-bp dizileri bulunmuştur ve çoğu montaj CHR üzerinde küçük dizilere sahiptir. 6 ve Chr. 8.
Ribozomal DNA (RDNA) 45S (18S, 5.8s ve 26s) ve 5S sekansları CBDRX/CS10 düzeneğinde (LOC115701787 5.8S, LOC115701759 5.8s, LOC115701762 26S ve Loc11572158 5s) ve pigenlere karşı kullanılır (Fignes. 1A ve genişletilmiş veriler Şekil. 5). İskele genomlarının karşısında 45S dizisi ağırlıklı olarak cHR’nin akrosantrik ucunda bulunuyordu. 8 ve 5S sadece Chr. 7 Kanabinoid sentaz kaset dizisi arasında, floresan ile yayınlanmış sonuçlarla tutarlı olarak, yerinde hibridizasyon106. Bununla birlikte, tüm kromozomlar üzerindeki bazı düzeneklerde kısmi diziler bulunmuştur (Genişletilmiş Veri Şekil. 5). Kısmi dizilerin farklı kromozomlar üzerindeki dağılımı genomlar boyunca değişkenliği yansıtabilir, çünkü bazıları montajlar arasında benzer yerleri paylaşır. Dizilerin çoğu, keçi alçalmasız kontiglerde bulunur, bu da farklı kromozomlar boyunca bu değişken dizilerin yanlış montajların sonucu olabileceğini düşündürmektedir. Genel olarak, esrar genomunda ortalama 1000 45 ve 2.000 5s dizisi vardır; Bazı montajlar 5S dizisini tamamen CHR üzerinde monte etti. 7.
VCF formatında genotip verileri107 VCFR kullanarak R’ye girdi108. VCFR ile alel ve heterozigot sayımlar yapıldı. Wright’s FKi hesaplandı109 Rastgele, Hardy -Weinberg, beklentimizden heterozigotlukta sapmayı sağlamak. Wright’s FKi (HS – HO)/HS olarak hesaplanmıştır, burada Ho, gözlemlenen heterozigot sayısının sayısı ve HS sayısıdır ve HS, ilk alel frekansı olarak hesaplanan alel frekanslarına dayanarak beklediğimiz heterozigotların sayısıdır. Dağılım grafikleri GGPLOT2 kullanılarak üretildi. Grafik paneller GGPUBR kullanılarak tek bir grafiğe monte edildi (https://cran.r-project.org/package=ggpubr).
Pankmer’ı kullanarak iki 31-mer dizinini oluşturduk: 193’lük bir ‘tam’ dizin Esrar Varsayılan parametrelerle ‘Pankmer Endeksi’ komutunu kullanarak düzenlemeler ve ‘Pankmer Endeksi’ komutunu kullanarak ‘sadece iskele sadece’ bir dizin. ‘Pankmer adj-matrix’ ve ardından ‘Pankmer Clustermap-Metric Jaccard’ kullanılarak tam indeksdeki tüm montajlar için çift Jaccard benzerliklerini hesapladık ve çizdik. Varsayılan parametrelerle ‘Pankmer Collection’ komutunu kullanarak hem tam hem de iskele sadece dizinler için bir toplayıcının eğrilerini hesapladık ve çizdik. Bu analiz için kullanılan tüm komut dosyaları GitHub’da bulunabilir.
Gen bazlı pangenomu, en az bir pangenom genomunda bir temsilci ile tüm gen aileleri (ortogruplar) kümesi olarak tanımlıyoruz. 193’ün her biri için (ve ayrı bir set olarak 78 kromozom düzeyinde, haplotip aşamalı genomlar) C. sativa Genomlar, her yüksek güvene bağlı gen tahmininin birincil transkripti temsilci olarak seçilmiştir. Her bir birincil transkript’e karşılık gelen proteinler, ortofinder kullanılarak ortogruplara kümelenmiştir (v.2.5.4, bkz. Aşağıdaki Orthofinder ve Synteny Analizi Bölümü)90. Birincil transkript CD’leri kümesi tek bir FASTA dosyasıyla birleştirildi ve kesin kopyalar Seqkit (2.7.0) ile çıkarıldı110. Birincil transkriptler arasında, olası kirleticiler, tahminlerin% 90’ından daha azının ‘viridiplantae’ veya ‘ökaryot’ olarak açıklandığı kontiglerde öngörülen transkriptlerin tanımlanmasıyla belirlenmiştir (v2.1.12)95ve kaldırıldı. Nesnelenmemiş genlerin problemini azaltmak için, tüm birincil transkriptlerin kodlama dizilerini 193 (78) esrar genomlarının her birine hizaladık (v2.26)75 Her genom için puro dizeleri içeren bir PAF dosyası oluşturmak için ‘minimap2 -c -x ek parametreleriyle. Her genom için, hizalanmış bir CDS sekansının en az 60 haritalama kalitesine sahip olması durumunda, sorgu uzunluğunun en az% 80’inde bir dizi puro eşleşmesi varsa ve doğrudan açıklanmış bir genin üst üste gelmediler, geliştirilmemiş bir gen olarak kabul edildi ve ortogroup hedef genomda mevcut olarak işaretlendi. 193 (78) genomun tümünde en az bir temsili olan ortogruplar kümesi çekirdek genom olarak kabul edildi, geri kalan ortogruplar değişken genom olarak kabul edildi. Her bir genomda her bir ortogroup’un varlığı veya yokluğu bir tabloda kaydedildi (bkz. Veri Kullanılabilirliği). Bu analiz için tüm komut dosyaları GitHub’dan edinilebilir.
Pangenomiklerde, koleksiyoncu eğrileri (pangenom nadirliği) haplotip sayısının ilişkisini gösterir (burada H) gen ailelerinin veya ortogrupların sayısına (burada X).
Göz önüne alındığında X Dağıtılan ortogruplar H haplotipler, skorun SX ∈ [0, H] bir ortogrup X içinde bulunan haplotip sayısı X mevcut. Herhangi bir puan için P(S) Skora eşit olan ortogrup sayısı olun S.
$$ p (s) = sum _ {x in {x} _ {0} … {x} _ {x}} {i} _ {{s} _ {x} = s} (x) $$ (x) $$
Nerede BENS_X: {X0…XX} → {0,1} {X ∈ X0…XX: SX= S}.
Koleksiyoncunun eğrisiC( H): [1, H] → [0, X] bir alt kümede bulunacak beklenen ortogrup sayısıdır. H Toplam kümeden rastgele çizilen haplotipler H. Şu şekilde hesaplanabilir:
$$ c (h) = sum _ {s in 1 … h} 1-p (s) prod _ {i in 0 in … h-1} frac {hsi} {hi} $$
Beklenen çekirdek ortogrup sayısı ({C}^{ wedge} (h) ) tarafından tahmin edilebilir
$$ {c}^{ wedge} (h) = sum _ {s in { rm {1 ..}}.
Bunların her biri, en azından puanla beklenen ortogrup sayısı için genel bir formül vakasıdır. Nhipergeometrik sağkalım fonksiyonuna dayanarak:
$$ {c} _ {n} (h) = sum _ {s in 1 … h} p (s) {s} _ {{hip}} (n, h, s, h) $$
Nerede Shiper hipergeometrik sağkalım fonksiyonu veya hipergeometrik kümülatif dağılım fonksiyonu 1’den çıkarılmıştır:
$$ {s} _ {{ rm {hip}}} (n, h, s, h) = 1-{{ rm {cdf}}} _ { rm {hip}}} (n, h, s, h) $$ $$
Netlik için, hipergeometrik olasılık kütle fonksiyonu (PMF):
$$ {{rm {pmf}}} _ {{ rm {hip}}} (n, h, s, h) = frac { left ( begin} {c} n end {array} sağ) , sol ( begin ( begin ( begin ( begin ( begin ( begin ( hn end {array} right)} { left ( begin {array} {c} h \ h end {array} right)} $$
Şu şekilde tanımlanan binom katsayıları ile:
$$ ( begin {array} {c} h \ n end {array}) = frac {h!} {n ,! (hn)!} $$
Ve geleneksel olarak, kümülatif dağılım fonksiyonu (CDFhiper):
$$ {{ rm {cdf}}} _ {{ rm {hip}}} (n, h, s, h) = sum _ {{n} n} {{ rm {pmf}}} _ {{ rm {hip}}} ({n} _ {i}, h, s, h) $$
Bu şekilde tanımlanmış, pan-genom koleksiyoncunun eğrisininC( H) eşdeğerdirC1( H), çekirdek genom koleksiyoncunun eğrisi ({C}^{ wedge} (h) ) eşdeğerCH( H):
$$ {c}^{ wedge} (h) = {c} _ {h} (h) $$
Toplayıcı eğrisinin tanımı, genomik sekans birimine agnostiktir, bu nedenle bir K-mer bazlı eğri, ortogrup bazlı eğri ile aynıdır, bu hariç X sayısı olacak K-Mers ve X Bir temsil edecek K-mer, bir ortogrup yerine.
Trim_galore, Ren ve ark.2 Kullanma: –2 Renk 2063. Bu okumalar daha sonra düşük bolluk okumaları için filtrelendi (trim -low -ibund.py -c 10 -m 5e9) ve daha sonra bir K-Mer Sketch (Sourmash Sketch DNA -P ölçekli = 1000, k = 31)79. Tüm pangenom düzenekleri de 31 -Mer frekansları açısından analiz edildi (Sourmash Sketch DNA -P ölçekli = 1000, k = 31). Son olarak, tüm Illumina Read ve Pangenome montajlarının tüm ikili örnekleri karşılaştırıldı (Sourmash -p 64 *.Sig -k 31). 31-mer mesafeleri daha sonra R’de (HCLUST (DIST (Sourmash_comp_Matrix), Method = “Ortalama”)) kullanılarak çizildi.
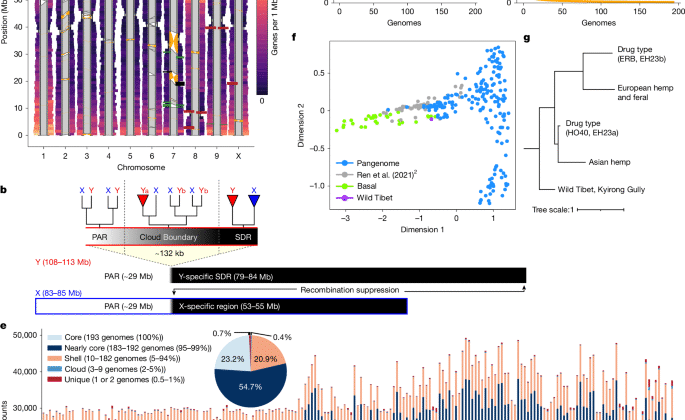
Ortogrup üyeliğine dayanan çekirdek ve dağıtılabilir (neredeyse çekirdek, bulut, kabuk, özel) genler atadık (https://github.com/padgittl/cannabispangenomeanalyses/tree/main/coredispensablegenes). Core genes were defined as being present in 100% of genomes (193 genomes), nearly-core genes were defined as being present in 95–99% of genomes (183–192 genomes), shell genes were found in 5–94% of genomes (10–182 genomes), cloud genes were found in 2–5% of genomes (3–9 genomes), and unique genes were found in Genomların% 0.5-1’i (1-2 genom)111. Bu analiz 193 genomun tümü üzerinde gerçekleştirildi (Şek. 1e) ve ayrıca popülasyona göre görselleştirildi (Ek Şek. 5). Beklenti seviyesi düzenekler (103 genom) için, sadece EH23A’nın on kromozomuna benzerliğe sahip contig’ler dahil edildi. Gen setleri, sadece on kromozomda mevcut olan ve kromozomlara homolog olan genleri içerecek şekilde süzüldü. Topgo ile fonksiyonel zenginleştirmenin bir analizini gerçekleştirdik73 Arka plan gen setinin belirli bir genom için bir GO terimi olan genler olduğu her genom için çekirdek, kabuk, bulut, neredeyse çekirdek ve benzersiz gen gruplamalarının her biri için. Çekirdek genler arasında, pangenomdaki en yaygın anlamlı GO terimi seskiterpen biyosentetik süreçti (Git: 0051762bir genom (PBBK) hariç tümünde önemli olan, ardından Git: 0045338 Üç genomda (kamu genomları: CAN, FIN ve PBBK) bulunmayan farnesil difosfat metabolik süreci (Ek Tablo 4). Bu analiz, Tsebra boru hattı ile öngörülen yüksek güven gen modelleri ile sınırlıydı. Aksine, koleksiyoncunun gen içeriğinin eğrisi analizi, gen modeli tahminlerinden yoksun, ancak bilinen genlere benzerlik ile örneklenmemiş çeşitliliği yakalamanın bir yolu olarak da dahil edilmemiş genom bölgelerini de içeriyordu (Şek. 1C, D ve Ek Şek. 4; ayrıca bkz. ‘Gen bazlı pangenomun analizi’).
Gösterilen ıraksama süresi tahminleri (Şek. 2b, c) denklem kullanılarak hesaplandıT= (1 – kimlik)/2Mdaha önce tarif edilen EDTA çıkışı GFF3 dosyalarından kimliğin elde edildiği yer97. Bir ikame oranı kullandık ( M) 6.1 × 10−9 itibaren Arabidopsis112–113. Bu analiz tüm genomlar üzerinde yapıldı.
Solo LTR’leri ve sağlam LTR-RT’leri tanımlamak için, EDTA boru hattını 193 esrar genomunda kullandık97. Teanno.gff3 dosyasının öznitelik sütununda ‘Method = homoloji’ temelinde alınan sağlam LTR-RTS olarak atanmayan LTRS kümesini ilk olarak toplayarak solo LTR’leri belirledik. Solo LTR’leri kesik ve bozulmamış LTR’lerden ve LTR-RT’lerin iç dizilerinden izole etmek için eşikler uyguladık. Bu eşikler, Referans LTR’ye göre minimum 100 bp, 0.8 kimlik ve minimum hizalama puanı içerir.114 300’ün. Bitişik LTR-RT ek açıklamasının aynı LTR-RT kimliğine sahip olmaması gerekir.115. Ayrıca, en yakın bitişik solo-ltr, sağlam LTR veya dahili diziye minimum 5.000 bp mesafe istedik116. Son olarak, LTR uzunlukları için 95. persentil içinde düşen Solo-LTR dizilerini sakladık117. Genel olarak, bu yöntem LTR_RETRIEVER’dan Solo_finder.pl komut dosyasına dayanan değiştirilmiş bir yaklaşımı temsil eder.114 ve ltr_miner betiği116 LTR_RETRIEVER için GitHub sayfasından rehberlik ile (https://github.com/oushujun/ltr_retriever/issues/41).
Plantenrikmen’in bir parçası olarak sunulan yöntem118 kannabinoid sentaz genleri de dahil olmak üzere farklı genomik özelliklerin hem akış yukarı hem de aşağı akışında TE zenginleştirmesini değerlendirmek için esrar pangenomu için uyarlanmıştır. Analizin amacı, belirli bir genomik özellik kategorisi ile önemli ölçüde ilişkili olan TE’leri tanımlamaktı. Kısacası, ‘x’ belirli bir TE türünü temsil eder ve ‘y’ tüm TE’leri kapsar. Belirli bir genomik özelliğin (örneğin, kannabinoid sentazların) yukarı akış veya aşağı akışlı toplam X sayısı A; Tüm genomik özelliklerin (örneğin, tüm genler) yukarı akış veya aşağı akışta bulunan toplam X sayısı B; Belirli bir genomik özelliğin (kannabinoid sentazların) yukarı veya aşağı akışında bulunan toplam Y sayısı C; ve tüm genomik özelliklerin (tüm genler) yukarı akış veya aşağı akışta bulunan toplam y sayısı D . Bir zenginleştirme skoru (ES) ({ rm {es}} = (a/b)/(c/d) )ve P Değer olarak tanımlanır (P = (A+B)! (C+D)! (A+C)! (B+D)!/(A! B! C! D!) ) )Neresi Ntoplamı A– B – CVe D . Çoklu test düzeltmesi119 üzerinde gerçekleştirildi P Python kütüphanesi statsmodels kullanan değerler120. Önem eşiği kesintileri, yanlış bir keşif oranı (FDR) <0.05 ve ES ≥ 2 içeriyordu.121 Genomik özellik kategorisinin 1 KB yukarı akışında veya aşağı akış katında bulunan TE setini toplamak ve incelemek. Örnek bir komut: bedtools kesişti -a montajıD_Genomic_Feature_coord_file.txt -b constblyId.te.gff3 -wo> AssectblyId_intersect_results.txt.
Genler ve TE kategorilerinin her biri arasındaki medyan ve ortalama mesafeler, bedtools sırası kullanılarak hesaplandı (bedtools sırası -i genome.tes.bed> genom.sorted.tes.bed) ve en yakın özellikler (komuta: en yakın-features –closest-header –dist genom.sort.genes.bed genom.sord.cles.122. Genler için önceden seçilmiş yatak dosyasını elde etmek için aşağıdaki komut kullanıldı: Cat Genes.gff3 | grep mRNA | grep ‘ .chr’ | Awk ‘{Yazdır $ 1 ” t” $ 4 ” t” $ 5 ” t” $ 7 ” t” $ 3 ” t” $ 9}’> genom.genes.bed. TES için aşağıdaki komut kullanıldı: Cat genom.edta.teanno.gff3 | grep ‘ .chr’ | awk ‘{yazdır $ 1 ” t” $ 4 ” t” $ 5 ” t” $ 7 ” t” $ 3 ” t” $ 9}’> genom.tes.bed. Ortalama ve medyan değerleri hesaplamak için yerleşik Python istatistik modülü kullanıldı.
Tam pangenomda farklı TE tiplerine yakın istatistiksel olarak konumda bulunan genleri tanımlamak için bir Go terimi zenginleştirme analizi gerçekleştirdik. TES yakınlarındaki genleri tanımlamak için, ilk olarak belirli bir gen için en yakın TE’yi bulmak için hem gen hem de TE koordinatları ile birleştirilmiş, sıralı bir yatak dosyası oluşturduk, ancak belirli bir gen için en yakın genomik özelliğin başka bir gen olduğu durumlar hariç. İskele genomları için, genler ve TE’ler on kromozom ile sınırlandırılmıştır. Beklenti seviyesi düzenekler için, on EH23A kromozomundan birine benzer bir kontig üzerinde olsaydı genler dahil edildi. Daha sonra, Bedops’un en yakın özelliklerini kullanarak gen/te çiftlerini belirledik122. Her genom için parametreler algoritması = ‘ağırlık01’, istatistik = ‘fisher’ ve Benjamini – Hochberg FDR ile çoklu test düzeltmesi ile her bir genom için ayrı ayrı bir Go zenginleştirme testi gerçekleştirdik.73. İstatistiksel karşılaştırma için arka plan gen evreni, belirli bir genom için bir GO terimi olan tüm genlerin kümesiydi. Geniş kalıpları değerlendirmek için, sadece en az beş genomda önemli olan GO terimleri daha fazla düşünülmüştür. Bu analiz tüm genom setini içeriyordu (Ek Tablo 11).
Kopyalarını çevreleyen 2 kb kuşatma mesafesi için genomik koordinatlar CBCAS, CBDA’lar Ve Thcas78 iskele için bedtools kanatları ile alındı (bedtools kanat -i montajıD_synthase_coords.bed -g chromizes.txt -l 2000 -r 2000> AssemblyId_flanking_2000.bed). Daha sonra, bu kuşatma bölgesinde yer alan TE’ler, Bedtools kesişme (bedtools kesişmiş -a constblyId_flanking_2000.bed -b constblyId.edta.teanno.gff3 -wo> constlicyId_intersect_2000.bed) kullanılarak alındı.121. Bedtools ile tanımlanan TE tiplerinin her biri için genomik diziler, bir fasta dosyasında toplandı ve mafft (mafft –auto helitron.fastta> helitron_aln.fasta) ile hizalandı.107. Fasttree (fasttree -nt -gtr -gamma helitron_aln.fastta> helitron_aln.tree) ile maksimum olabilirlik ağacı inşa edildi)123. Ağaç figtree ile görüntülendi124. Tüm LTRS kümesinde fazlalığı azaltmak için, çoklu dizi hizalamasından önce diziler kümesine CD -HIT uygulandı (CD -HIT -EST -i ty1_ltrs.fast.125.
EDTA’nın yedek olmayan TE dizisi kütüphanesi ‘transkriptom’ olarak somona sağlandı. EH23 RNA-seq numunelerinin her biri TE transkriptomuna eşlenmiştir. Gen ekspresyon analizine benzer şekilde, belirli bir TE için minimum TPM eşiği, numunelerin ≥% 20’sinde ≥0.1 tpm idi126. İfade edilen en iyi 50 TE, bir ısı haritası olarak görüntülendi, günlüğü gösteriyor2TPM log kat değişimini temsil etmek için.
‘CPG Adaları’> 200 bp, GC içeriği>% 50 ve gözlemlenen/beklenen CPG oranı> 0.6’yı kapsayan metillenmemiş bölgeler olarak tanımlanır. Zaman içinde sitozin metilasyonu, sitozin timine deamine edildikten sonra CpG dinükleotit kaybına neden olur. Sitozin metilasyonu ile beklenti, CPG dinükleotitlerinin (CG, CHG, CHH (burada H A, T veya C’dir)) daha fazla metilasyon aktivitesine sahip olacağıdır. Gözlemlenen/beklenen CPG oranı hesaplaması127–128 :: ({ rm {cpg}} , { rm { rm { rm {count}}/l)/( rm {c}} , { rm {count}}} , { rm { rm { rm {cot {{ rm {count { rm { rm {count { rm { rm { rm { rm { rm { rm { rm { rm { rm}. Gözlemlenen/beklenen CPG modelleri Şek. 2H, K.
SV alt tiplerinin her biri (inversiyonlar (Inv), kopyalar (DUP’lar), translokasyonlar (trans) ve ters translokasyonlar (InvTR)), her bir kesme noktasının (her bir kırılma noktası için toplam 1 kb yukarı akış ve aşağı akış bölgesi (her bir kesme noktası için toplam 1 kb), hem sağlam hem de parçalanmış ek ekleme kullanılarak TE içeriği kullanılarak incelenmiştir. Nüfus tarafından gruplandırılmış 78 iskele, kromozom seviyesi genom seti dahil edildi. Genom ile karşılaştırmak için, aynı genom ve kromozomdan rastgele bir pencere, Bedtools karışıklığı olan SV’lerin her biriyle aynı uzunlukta alındı ve kuşatma pencereleri simüle edilmiş kesme noktalarının her biri için alındı. Sadece belirli bir TE tipinin tek bir SV’nin her iki kesme noktasıyla ilişkili olduğu durumlar, ayrıca bedtools kesişmesiyle değerlendirildi. Bu analize hem parçalanmış hem de sağlam TE’ler dahil edildi. İstatistiksel önem, Welch’in iki taraflı kullanılarak değerlendirildi T-Scipy’de testi71. TE’ler, aynı kromozom ve genomdan aynı uzunlukta rastgele seçilen bölgelere göre SV kesme noktalarına (kesme noktasının yukarı ve aşağı akışta 500 bp; toplam 1 kb) daha sık görülür. Bolluktaki farklılıkların üstesinden gelmek için, genomun rastgele karıştırılmış bölgeleri bootstrapped (1.000 kopya), simüle edilmiş, karıştırılmış TE veri kümelerinin her birinin popülasyonda gözlemlenen kesme noktalarının sayısıyla eşleşmesi gerekliliği. Gözlemlenen ve simüle edilmiş verilerin TE içeriği, Welch’in iki taraflı ile istatistiksel anlamlılık açısından değerlendirildi. T -Scipy’de testi71 ve Benjamini – Hochberg Çoklu Test Düzeltmesi (alfa = 0.5, yöntem = ‘bağımsız’, is_orted = false)120. Bir test istatistiği ve P 1000 bootstrap kopyasının her biri için değer üretildi. Ortalama test istatistiği ve PDeğer daha sonra hesaplandı (Ek Tablo 13).
193 esrar proteomunun analizine yardımcı olmak için ortofinder sürüm 2.5.4’ü çalıştırdık. İki çalışma tamamlandı. Birincisi, en kaliteli esrar montajlarımıza odaklandı ve sadece iskeleli montajları ve Plaza’dan düzinelerce diğer bitki örneği ve NCBI’dan birkaç örnek içeriyordu. Kalan montajların ayrıntılı protein seviyesi analizine izin vermek için Plaza’dan kaynaklanan yakın akrabalarla birlikte tüm esrar pangenom montajlarımız da dahil olmak üzere bir başka çalışma da üretildi. Tüm durumlarda, sadece birincil (aksi halde açıklanmadıkça en uzun izoform) protein sekansı kullanıldı. OrthoFinder sonuçları, ortobrowser dahil olmak üzere çeşitli yöntemler kullanılarak analiz edildi.129gen ağacı dendrogramlarının eşzamanlı görselleştirilmesine, gen ağacı çoklu dizi hizalamalarına ve seçilen gen ve çevresindeki genlerin tüm genomlar boyunca sintenyasına izin veren statik web sayfaları üretebilen (https://resources.michael.salk.edu/root/home.html).
İskele esrar ortofinder çalışmasına dahil olan kanal dışı genomlar: (1) Amborella Trichopoda; (2) Aquilegia Oxysepala; (3) A. thaliana; (4) C. sativa; (5) Carpinus Birkaç; (6) Carya Illinoinensis; (7) Ceratophyllum Demersum; (8) Citrullus Lanatus; (9) Corylus Avellana; (10) Salatalık melo; (11) Cucumis sativus; (12) Taze Parçalar; (13) Fragaria x; (14) Lotus japonicus; (15) Bondiy manolya; (16) Malus domestica; (17) Manihot escultenta; (18) M. Dikkate değer; (19) Zamir; (20) Oryza sativa; (21) Parasonia Anderson; (22) A. Şeftali; (23) Quercus lobata; (24) Rosa Chinensis; (25) Başarıya Sechium; (26) T. Orientale; (27) Trochodendron Aralioides; (28) Vaccinium Macrocarpon; (29) V. Vinifera; (30) Z. Jujuba; ve (31) H. Lupulus.
Tam Esrar Orthofinder Run’da bulunan kanal dışı genomlar: (1) F. Flex; (2) L. japonicus; (3) M. Dışa; (4) A. Şeftali; ve (5) R. Chinensis .
193 genomda protein ve DNA bazlı ortogruplar için sekans entropisini hesapladık. Yüksek entropi, bir ortogruptaki sekanslar arasında daha fazla çeşitliliğe ve varyasyona karşılık gelir ve düşük entropi, ortogrup dizileri arasında daha az çeşitliliği ve daha fazla benzerliği gösterir. Minimum 0 entropi değeri eşleşen kimliğe karşılık gelir. Maksimum entropi rastgele bir amino asit dizisine karşılık gelir ve denklemden türetilir: log2(20) = 4.32, burada 20 amino asit sayısıdır. DNA için maksimum entropi130 günlük2(4) = 2.0. SciPy.stats’tan entropi işlevini kullanarak ortofinder çoklu dizi hizalamasının her sütunu için entropiyi hesapladık.71 ve daha sonra tüm çoklu sekans hizalaması için ortalama entropi hesapladı. Analize dahil edilmek için ortogrup başına en az beş sekans gerekiyordu. Popülasyonlar boyunca her ortogrup için ikili karşılaştırmalar yapıldı ve her çok sekans hizalaması için entropi değerlerinin dağılımı eklem histogramı olarak görüntülendi. Bu analiz hem proteinlere (gen dizileri) hem de DNA’ya (TES) uygulandı.
Haplotip çözünmüş, kromozom ölçeğinde genomlardaki gen seviyesi varyasyonunu X ve Y kromozomları (AH3M, BCM, GRM ve Komp) ile görsel olarak değerlendirmek için Genespace sürüm 0.9.3 kullandık.131 R Sürüm 4.2.2 (2022-10-31)132. Başlangıçta Orthofinder’ı çalıştırdık90 Genespace ortamının dışında ve sonuçları içe aktardı. Analizi çalıştırmak için Synteny işlevini, ardından Plot_Riparianhits’i kullandık. Pangenom fonksiyonu ile bir pangenom temsili oluşturduk. Alt kümedeki tüm genomlarda sintenik gen çiftleri elde etmek için kullanılan birincil dosya olarak gffwithogs.txt çıktı dosyasını kullandık. ‘OG’ sütununda (son sütun) özdeş tamsayı değerine sahip gen kimlikleri sintenik ortologlar olarak alındı.
78 tamamen iskele montaj haplotiplerinin her biri, minimap2 kullanılarak EH23A montajına hizalandı75. Syri daha sonra her hizalamada SVS’yi aramak için kullanıldı98 ve hizalamaları ve SV’leri görselleştirmek için PLOTSR kullanıldı99. CD’ler ve TE içeriği, Bedtools kesişerek analiz edildi121. İnversiyon kırılma noktası tekrarları, minimum 10 kb boyutunda inversiyonların blastn hizalamaları kullanılarak çağrıldı. 8 kb’lik pencereler, her bir ters çevirmenin başlangıç ve uç kesme noktasının etrafında ortalanmış ve kendiliğinden kendiliğinden hizalanmıştır ve ters çevirmenin karşıt tarafındaki kırılma noktası pencere çifti (baştan sona). Kesme noktası başına sadece en iyi puanlama hizalaması (tam uzunlukta benlik-benlik hizalaması hariç) sayıldı. Tersine çevrilmiş tekrarlar, zıt yönelimlerde hizalamalar olarak adlandırıldı ve segmental kopyalar aynı yönde hizalamalar için çağrıldı.
SNP’lere Syri kullanılarak da adlandırıldı98 Yukarıda açıklandığı gibi aynı montajlarda ve hizalamalarda. Örnek başına iki haplotipin her birinden SNP’ler, numune başına tek aşamalı genotip çağrılarıyla birleştirildi ve alt çağrısı çıkarıldı (github.com/rclynch414/syri_vcf.sh). Son olarak, VCftools kalite filtrelemek ve ince SNP alanlarını minimum 1000 bp aralığına kadar kullanıldı: —semove-indels —mingq 20 —semove-indv eh23a–din-alleles 2 —max-aleles 2-1000 —stdout —Recode.
İskele montajlarından gelen aşamalı SNP’ler önce plink kullanılarak çöp kutusu ile R2 korelasyonları açısından değerlendirildi133: —double-id –wall-extra-chr —set-missing-var-ids @:# —maf 0.01-Geno 0.1-Mind 0.5 –Chr 7 –thin 0.1 -r2 gz –ld-window 100 –ld-window-kb 1000 –ld-window-r2 0-Make. Daha sonra LD_DECAY.PY, R’de ggplot ile çizilen bozunma eğrileri (GitHub -Erikrfunk/genomics_tools) yapmak için kullanıldı. ve r (sfustatgen.github.io/ldheatmap/) ldheatmap ile çizildi.
GO terim zenginleştirme testleri, EH23A’nın null dağılımı ve klasik Fisher testi olarak EH23A’dan gelen tüm yüksek güven gen ek açıklamaları kullanılarak R’deki Topgo paketi ile gerçekleştirildi.73.
FST Değerler, her aşamalı SNP ve iskele montaj MJ ve kenevir popülasyonu atamaları için VCFTools kullanılarak hesaplandı; Anlamlılık, bu değerlerin ilk% 5’i kullanılarak hesaplanmıştır. Seçici süpürmeler için XP-CLR modeli, aynı SNP’lere ve 20 kb genom dulları 59’a uygulandı; Anlamlılık, bu değerlerin ilk% 5’i kullanılarak hesaplanmıştır.
TreeMix modeli sadece gen modellerinin dışında SNP’ler kullanılarak çalıştırıldı: -Eed 69696969 -o out_stem -m 5 -k 50 -noss -noss -root Asian_Hemp. Bir ila 10 göç senaryosu simüle edildi ve LN’ye (olasılıklar) göre sıralandı. Beş göç olayı (-m = 5) en olası nihai sayı olarak seçilmiştir.
Yerel PCA yöntemi, aşamalı SNP’lere uygulandı, SNP’ler arasında minimum 1.000-bp aralık ve 100 SNP’nin genom pencereleri134.
Bitki hastalığı direnci gen analogları, kodlanmış proteinlerinde bir veya daha fazla yüksek oranda korunmuş amino asit motiflerinin varlığı ile tanımlanır. Bu motifler, patojen özgüllüğünü ve hücre altı lokalizasyonunu belirleyen fonksiyonel protein alanlarını kodlar. Belirli patosisteme bağlı olarak, direnç gen analog proteinleri tamamen sitoplazmik olabilir veya hücre zarını sitoplazmik fonksiyonel alanlar, hücre dışı alanlar veya her ikisi ile yayabilir.
Drago2135 78 kromozom düzeyinde, haplotip çözünmüş genomlar için bitki hastalığı direnç gen analogları arasında korunan motifleri tanımlamak için kullanıldı. Giriş dosyaları, her genom için transkript ek açıklama fasta dosyalarıydı. Hem nükleotid bağlanma bölgesi (NBS) hem de lösin açısından zengin tekrar (LRR) alanları içeren gen setleri, gen setleri üzerindeki motiflerdeki amino asit bileşimini değerlendirmek ve karşılaştırmak için meme için girdi olarak kullanıldı.
Külce direnci ile ilgili genleri tanımlamak için, Chr ile eşlenmiş bir markerin dizisi. CBDRX’te 2, EH23A ankraj genomuna karşı Blastn sorgusu olarak kullanıldı136. Ortaya çıkan isabet, CHR’de% 96 nükleotit kimliğine sahipti. EH23A’nın 2’si 77,292.037-77.291.397 bp. 32 kinaz alanları, altı reseptör benzeri kinaz, ikisi nükleotit bağlanma bölgesi artı transmembran alanları, biri sarmal bobin ve kinaz alanları ile ve diğeri sarılı bobin, nükleotit bağlanma bölgesi ve transmbran alanları içeren 46 gen kümesinde yer alıyordu. Blast vuruşu, iki açıklamalı kinaz geni, EH23A.CHR2.v1.g115480 ve eh23a.chr2.v1.g115510 arasındaydı.
Ortaya çıkan üst patlama hitleri herhangi bir gen ek açıklaması ile örtüşmedi; Bununla birlikte, 38 genomun 16’sının Chr. 2 CBDRX genine>% 95 nükleotit kimliği ile; Bunlardan dokuzu, üç eksonun hepsinde (1.745 bp, 1.448 bp ve 287 bp)% 99-100 nükleotit kimliğine sahipti. 16 genomun beşinden (H3S7A, OFBA, SZFBA, TKFBA ve WCFBA) sekanslar, diğerlerinden ayrı olarak kümelenmiştir. Bunlar, birinci eksona 1-bp yerleştirme, eksonik uzayda on küçük indel (2-8 bp) ve 1.280 bp daha uzun ikinci intron ile ayırt edildi. Bu bölgeler çıkarıldı ve CBDRX gen dizisi ile hizalandı ve hizalama, maksimum olabilirlik ağacı üretmek için kullanıldı (Genişletilmiş Veri Şekil. 8).
Sarmal-bobin NBS-LRR genleri (CNL’ler) CHR üzerinde farklı bir desen gösterdi. 3 ve Chr. 6. 400-600 kb arasında bir ila iki CNL geni vardı; 1-1.4 MB arasında iki ila dört; 6-8 MB’de bir ila iki; 35-37 mb’de kromozomun sentromerik bölgesine yakın tek bir CNL geni ve 78-84 MB arasında bir ila beş (COFBA) CNL’ler. Bu modelin istisnaları, sentromerik bölgede bir CNL eksikliği olan OFBA, H3S1A ve MMV31A idi. SDFBA ve SN1V3A’da, sentromerik CNL’ler sırasıyla 42.8 ve 47.5 Mb’de bulundu. SN1V3A’nın 12.2 MB’de bir CNL vardı, genel desen için bir başka istisna vardı. Chr. Bu genomda 3, diğerlerinden daha büyüktü, 90 mb’de, geri kalanına 80-85 MB. Son olarak, Gerv1a, CHR’nin 78-84 MB bölgesinde bir CNL yoktu. 3.
Her biri Esrar Proteomlar, BLASTP (Sürüm Blast 2.6.0, Build 7 Aralık 2016) ile UniProt (‘Embryophyta’ ve ‘İncelenen’; 20 Eylül 2022’de erişilen arama kriterlerinden 40.926 protein dizisine hizalandı.137. Hizalama eşikleri, 10’dan az bir e-değeri eşiği içeriyordu−3en az% 20 sorgu kapsamı ve hizalamanın uzunluğuna dayanan yüzde bir kimlik138. Terpen sentazları ayrıca PFAM alanlarının varlığına, PF01397 ve/veya PF03936139. Etki alanı içeriğini değerlendirmek için her biri Esrar Proteomlar PFAM-A.HMM veritabanına hizalandı (son değiştirildi 15 Kasım 2021; erişim 20 Eylül 2022)140 HMMSCAN ile (HMMER 3.3.2 Kasım 2020)141 varsayılan ayarlarda.
Terpen biyosentezi iki yol yoluyla ilerler: kloroplastik metil-D-Monoterpen ve kannabinoid biyosentezi için öncüler ve seskiterpen biyosentezi için öncüler üreten sitosolik mevalonat yolu üreten -eritritol fosfat yolu. Bu yollar için protein dizileri142–143–144 her birine hizalanmış Esrar Varsayılan ayarlarda elmas sürüm 2.1.4’ü olan proteomlar145.
193 kenevir genomlarının her birinde tam ve kısmi uzunluk kannabinoid sentazlarını tanımlamak için, referans kanabinoid sentaz sekansları Blastn ile genomla hizalandı. CBDRX’ten geliştirilen zenginleştirilmiş bir LTR dizisi11 sentazların tanımlanmasında daha fazla yardım için referans olarak kullanılmıştır. LTR08, CBDRX genomundan sentaz kasetleri ile ilişkili bir LTR dizisidir. Cannabinoid sentaz patlama sonuçları ve LTR08 patlama sonuçlarını tablo formatında almak için bir python betiği yazılmıştır. <500 bp uzunluğunda sentaz isabetleri filtrelendi. Bitscore <1.250 ile LTR08 isabetleri filtrelendi. Synthase ve LTR08, <10 ve sıfır boşluklarla uyumsuzluklarla isabetler 'tam' diziler olarak etiketlendi. Diğer tüm isabetler 'kısmi' diziler olarak etiketlendi. Aynı başlangıç pozisyonunu paylaşan isabetler daha sonra tek bir diziye süzüldü ve aşağıdakilere göre sentaz etiketlerinden biri verildi. Tam isabetler korundu ve karşılık gelen fonksiyonel sentaz olarak etiketlendi. Yukarı akış veya aşağı akışta bir LTR08 vuruşunun 60 KB'si içinde kısmi isabetler CBDA’lar ve tutuldu. Yakınlıkta bir LTR08 ile tam hit veya isabet yoksa, en yüksek bitscore ile vuruş ilgili sentaz olarak etiketlendi ve tutuldu. Filtrelenmiş ve etiketlenmiş sentazlar daha sonra bir genomun her bölgesi için kannabinoid sentaz oryantasyonunu görselleştirmek için bir iz üzerine çizildi. Görselleştirme için en az dört sentaz isabetine ihtiyaç vardı. Inkscape, sentaz kaset izlerini görselleştirmek için kullanıldı. Manuel düzenlemeler, birkaç yanlış etiketi düzeltmek için kullanıldı. CBDA’larVe CBCA’lar. Synthase kasetleri genel kaset şekli ile gruplandırılır.
İlk Orfinder, psödojenleri yukarıda tarif edilen potansiyel genlerin ilk listesinden çıkarmak için kullanıldı (ftp.ncbi.nlm.nih.gov/genomes/tools/orffinder/linux-i64/). Sonra Usearch11.0.667’yi küme sentaz kodlama dizilerini kullandık: -cluster_fast -id -id 0.997 -Sort uzunluğu -d her iki -cenroid -kümeler146. Daha sonra tercüman, protein güdümlü çoklu sekans hizalamaları üretmek için kullanıldı147. Synthase evrimsel tarihi, Mega11’de maksimum olabilirlik yöntemi ve genel zaman geri dönüşümlü model kullanılarak çıkarıldı.148.
Pankmer’in ankraj fonksiyonunu, bilinen esrar genotiplerinde crossover olaylarını bulmak için kullandık (Ek Tablo 15). On bir trios, Varin-Donor ebeveyn olarak FB191’i ve 6 üçlüsünün Varin-Donor ebeveyn olarak SSV’yi içerdiğini içeriyordu. FB191’in ebeveynleri HO40 ve FB30 iken SSV’nin ebeveynleri HO40 ve SSLR’dir; Her iki durumda da, Ho40 Varin donörüydü. Her üçlü için f1 Genom haplotiple çözüldü ve bir varin-donör ebeveyninden bir haplotip ve bir Varin olmayan donör ebeveynden bir haplotip içeriyordu. Her durumda, ‘Varin haplotipini’ tanımlamak için Pankmer demirleme kullandık. FB191 üçlüsü için, varsayılan parametrelerle ‘pankmer endeksi’ kullanarak FB191 genomunun 31-mer indeksini oluşturduk. Python komut dosyası kullanma Pankmer’ın API işlevlerini içe aktarma Pankmer.anchor_region () ve pankmer.anchor_genome ()21örneğin COFBA ve COFBB gibi haçın her haplotipine FB191 endeksini demirledik. Varin haplotipini FB191 endeksinde 31-mer korumasına sahip haplotip olarak tanımladık. Aynı prosedürü SSV’nin bir pankmer endeksi kullanarak SSV Trios’a uyguladık. Daha sonra potansiyel Varin alellerini HO40’dan haçın varin haplotipine kadar izlemeye çalıştık. HO40’ı temsil etmek için, iki tek nesil 31-Mer indeksleri ürettik: biri Ho40 genomu için ve diğeri de oldukça benzer EH23A sekansı için. Ayrıca FB30 ve SSLR’nin tek Genome 31-Mer indeksleri ürettik. Her FB191 haçı için Varin haplotipinde HO40 endeksini, EH23A dizinini ve FB30 dizinini sabitledik. Loci’deki crossover olaylarını net bir ‘haplotip anahtarı’ ile gösterdik. K -mer koruma değerleri. SSV Trios için aynı prosedürü tekrarladık ve SSLR endeksini FB30 endeksi yerine uyguladık. Bu analiz için tüm komut dosyaları GitLab’da mevcuttur.
İlk olarak, başlangıçta çok modal olarak kabul edilen varin oranı verilerini dönüştürmek için sıralı kantil (ORQ) yöntemini seçmek için R’deki en iyi normalize paket kullanıldı. Sonra model R’deki boşluk paketinden yanıp söner149 F’deki SNP’ler arasındaki ilişkileri test etmek için PCA.total = 6 ile kullanıldı2 popülasyon ve dönüştürülmüş varin oranı verileri (Ek Tablo 16). Bu PCA.total parametre, PCA için QQ grafiklerinin görsel değerlendirmesine dayanılarak seçildi. Pdeğer149. Daha sonra, FDR düzeltilmiş dört önemli SNP’yi çevreleyen bölgelerde gen ve TE modelleri manuel olarak değerlendirildi (Ek Tablo 16), K-Mer tabanlı çapraz sonuç. Dört önemli SNP’den, açıklanan en yüksek iki fenotipik varyansla ilişkili genler üzerinde daha fazla analiz odakladık (Ek Şek. 25). Sonra, için ortofinder grupları BKR – Alt3 Ve Alt4 çıkarıldı ve üçü Alt3 Ve Alt4 Ortogruplar tek bir alt gen sayımlarına yerleştirildi. BKR ve ALT protein dizilerinin filogenileri, 100 bootstrap replikatları kullanılarak ortogruplardan komşu birleştirme yöntemi ile mega içinde inşa edildi.148. . BKRGörüntülenen hizalama ve çeviri, Geneious kullanılarak yapıldı150 Varsayılan ayarlarda hizalama algoritması (Şek. 5).
Y tabanlı K-MERS (Y-merler), BWA (V.0.7.17) MEM kullanılarak X/Y haplotiplerine eşleştirildi, mükemmel hizalamalar gerektirdi ve 10 kez kadar multIpaping’e izin verdi. Varsayılan SDR-PAR sınırlarını belirlemek için, sonraki gen ağacı analizi için azalmış Y-mer haritalama yoğunluğuna sahip bölgelerde korunmuş ortologları çıkarmaya odaklandık. Orthologlar, çoklu dizi hizalama seçeneği ile ortofinder (v.2.5.4) kullanılarak tanımlandı. OrthoFinder, tüm erkek ve birkaç kadın kontig seviyesi montajları dahil olmak üzere bu çalışmadan mevcut tüm erkek (XY) montajlarından proteinler kullanılarak yürütüldü ve diğer çalışmalardan ilave haplotip çözülmüş montajlar: (1) boaxa; (2) boaxb; (3) AH3MA; (4) AH3MB; (5) BCMA; (6) BCMB; (7) GRMA; (8) BCMB; (9) GRMA; (10) carmagnola_hap229; (11) futura75_hap129; (12) futura75_hap229; (13) Ottoii_hap129; (14) Ottoii_hap229; (15) USO31_HAP129; (16) USO31_HAP229; (17) fimv1a; (18) fimv1b; (19) GVA-H-22-1061-002_HAP134; (20) GVA-H-22-1061-002_HAP234; (21) GVA-H-21-1003-002_HAP134; (22) GVA-H-21-1003-002_HAP234; (23) SAN2A; (24) SAN2B; (25) tibv1a; (26) TIBV1b; (27) WFV1A; (28) WFV1b; (29) WIV1A; (30) WIV1b; (31) YMMV1A; ve (32) YMMV1B.
Gen ağaçlarının, hangi ortologların SDR olduğunu veya her bir montajda par-bağlantılı olduğunu belirlemek için varsayılan SDR-PAR sınırlarını kapsayan on korunmuş ortolog için tahmin edildi. Örneğin, y gametologu (x ve y kromozomlarındaki 1: 1 ortologlar) SDR ile sıkı bir şekilde bağlandığında, X- veya Y’ye bağlı ortologlar içeren ayrı klapeler için güçlü destek beklenir.151.
Korunmuş on ortolog veya gametologun tümü için: (1) tam uzunlukta genler (intronlar dahil) için nükleotit sekanslarını bulmak ve çıkarmak için Blastn (Blast+ V.2.14.1) ve Bedtools (v.2.31.0) Getvasta kullandık; (2) ‘-localpair-Maxiterate 1000’ seçeneklerini kullanarak her bir gen matrisini mafft (v.7.505) ile hizaladı; ve (3) ‘-mfp -bb 1000’ seçenekleriyle IQ-Tree (v.1.6.12) ile maksimum olabilirlik ağaçları. X-Y gametolog ağaçları analizimizi takiben, her bir SDR sınırını tanımlamak için ilk varsayılan Y-spesifik, SDR bağlantılı gene karşılık gelen gen koordinatlarını kullandık, daha sonra yastıklı başlangıç koordinatlarını 10 bp. X’e özgü bölgelerin başlangıcı (yani x üzerindeki y ile yeniden birleşmeyen ve y-sdr ile kollinear olan bölge), ilk Y-spesifik gene karşılık gelen X-gametolog koordinatlarına dayanarak tanımlanmıştır.
SDR-PRAP SINIR, SDR sınırlama bölgelerinden XY gametologların gen ağaçları kullanılarak tanımlandı ve buK-Her haplotip için. Gen ağacı analizimiz, farklı SDR sınırlarına (YA ve YB) sahip iki ana y haplotip grubunu ortaya çıkarmıştır. ‘Bulut sınırı’, XY gametolog ilişkilerine dayanarak esrar içindeki SDR -Par sınırındaki varyasyonu temsil eder. Veri kümemizde daha yaygındı (N= 6) ve bulut sınırını kapsayan ~ 132 kb genişletilmiş bir SDR sergiler; oysa bu bölge daha az sıklıkta, yb, haplotipte paraya bağlı kalır (N= 2). Ana metinde bildirilen YA haplotipi, kuzey İtalya’dan bir lif kenevir landrace olan BCMB (Feral), GRMA (HC Kenevir), AH3MB (MJ) ve Carmagnola’da bulundu ve Yb haplotipi, 1950’de seçilen Kompolti’de (Macar fiber külti) seçildi, 1950’de seçildi ve 1950’de daha eski bir şekilde seçildi ve 1950’de daha eski bir şekilde seçildi ve 1950’de seçildi ve 1950’de seçildi ve 1950’de seçildi ve 1950’de seçildi ve 1950’de 1950’de seçildi. GVA-H-21-1003-002 (NY, ABD’den izole edilmiş vahşi nüfus).
Araştırma tasarımı hakkında daha fazla bilgi,Doğa portföyü raporlama özeti Bu makaleye bağlı.
 1
Küresel ısınma sayesinde toz fırtınaları için mükemmel fırtına
40 kez okundu
1
Küresel ısınma sayesinde toz fırtınaları için mükemmel fırtına
40 kez okundu
 2
Yok olan bir savanın ortasında, yeni koridor vahşi yaşam için ‘büyük bir kazanç’
15 kez okundu
2
Yok olan bir savanın ortasında, yeni koridor vahşi yaşam için ‘büyük bir kazanç’
15 kez okundu
 3
Küçük bir ada sürdürülebilirlikte büyük dersler sunar
14 kez okundu
3
Küçük bir ada sürdürülebilirlikte büyük dersler sunar
14 kez okundu
 4
Gen Z, ağaçlıklarla koruma çağrısına liderlik ediyor
13 kez okundu
4
Gen Z, ağaçlıklarla koruma çağrısına liderlik ediyor
13 kez okundu
 5
Hedefsiz varyasyonlar: Mayıs 2025
12 kez okundu
5
Hedefsiz varyasyonlar: Mayıs 2025
12 kez okundu